

NVIDIA SWI UNETR模型在医疗中的应用
描述
NVIDIA 数据科学家本周在享有盛誉的MICCAI 2021医学成像会议上,在脑肿瘤分割挑战验证阶段占据了前 10 名的三位。
现在已经进入第十个年头, BraTS 挑战项目要求申请者提交最先进的人工智能模型,用于在多参数磁共振成像( mpMRI )研究中分割异质性脑胶质母细胞瘤亚区域,这是一项极具挑战性的任务。
参与者还可以关注分类方法的第二项任务,以预测 MGMT 启动子甲基化状态。
2000 多个 AI 模型被提交给了挑战,该挑战由医学图像计算和计算机辅助干预学会、北美放射学会和美国神经放射学会联合组织。
NVIDIA 开发者在挑战验证阶段排名第一、第二和第七,每个人都创建了不同类型的用于肿瘤分割的 AI 模型方法,包括优化的 U-Net 模型、具有自动超参数优化的 SegResNet 模型和基于 transformer 的计算机视觉方法的 Swin-UNETR 模型。
NVIDIA 获奖者都使用了开源 PyTorch 框架MONAI(人工智能医疗开放网络),这是一个由学术界和行业领袖构建的免费、社区支持的计划,旨在将医疗成像深度学习的最佳实践标准化。
用于脑肿瘤分割的优化 U-Net –排名# 1
该优化 DU 网络模型是一种编码器 – 解码器类型的卷积网络体系结构,用于快速、精确的图像分割,它首先处于 BraTS 验证阶段。它的标准化统计排名得分为 0 。 267 。
设计优化 U 型网络的起点是 BraTS 2020 获奖解决方案:神经网络在脑肿瘤分割中的应用。该团队的目标是优化 U-Net 体系结构以及培训计划。为了找到最佳的模型结构,数据科学家进行了广泛的烧蚀研究,发现香草 U-Net 和深度监督产生了最好的结果。
进一步优化 U-Net 模型,在输入端添加额外通道,对前景体素进行一次热编码,将编码器深度和卷积通道数量增加一个级别。通过在较低的解码器级别上添加两个额外的输出头,可以更好地实现梯度流和更准确的预测,从而在深度监督下更好更快地训练模型。MONAI用于数据预处理,以清除数据并去除数据中的噪声,以及推断模型。 NVIDIA 数据加载库( DALI )使用并支持数据扩充,这是一种用于人为扩大数据集大小的技术,它通过将数据扩充卸载到 GPU 来解决 CPU 瓶颈问题。
带有最新 PyTorch、cuDNN 和 CUDA 版本的 NVIDIA PyTorch 容器用于优化 U-Net 模型以进行快速训练。自动混合精度 (AMP) 用于将 AI 模型的内存占用减少 2 倍并加快训练速度。训练在 8 个 NVIDIA A100 GPU 上完成 1000 个 epoch,与 V100 GPU 相比,速度提高了 2 倍。几乎 100% 的 GPU 被使用,展示了网络优化以高效使用 GPU。此 3D U-Net 模型可用于任何 3D 模式,例如 MRI 和 CT。了解更多 GPU 高效的 nnU-Net 实现。

图 1 。挑战验证数据集上的预测。在第一行, T2 模态是可视化的。第二行显示以下颜色的模型预测:紫色 – 背景、蓝色 -NCR 、绿松石色 -ED 、黄色 -ET 。
SegResNet : 3D 大脑磁共振成像语义分割中的冗余减少–排名# 2
该方法在 BRAT 挑战中排名第二,将在 MONAI 中提供,基于 MONAI 组件,旨在展示其应用的实用性和灵活性。主要模型是 MONAI 中的 SegResNet 架构,这是一种基于标准编码器 – 解码器的卷积神经网络( CNN ),类似于 U-Net 。该方法是 MONAI 自动化( AutoML )计划的一部分,使用超参数优化和调优自动选择超参数。
该方法在验证阶段的排行榜( team NVAUTO )中取得了最佳性能,并在基于个案排名和扰动分析的综合排名中取得了 0 。 272 的排名。组织者表示,该排名与第一排名的解决方案在统计上没有显著差异,两种方法被认为在统计上相似。
这项工作增加了两项新贡献,以进一步提高性能。首先,对训练过程进行修改,以在学习的特征表示上强制执行某些属性。通过借鉴自监督文献中的思想,将特征维度正则化,使不同解剖区域之间的冗余度最小。同时,相同解剖结构的区域被鼓励相似。这允许更好的网络行为和泛化。其次,采用自适应融合技术自适应地选择模型子集进行融合。这有助于避免某些模型预测中的潜在异常值,并进一步提高最终的集合性能。
该方法在 MONAI 中实现,以 PyTorch 为基础,并在四个 NVIDIA V100 GPU 上使用骰子损失函数进行了为期 16 小时的 300 个历元的训练。该团队使用 NVIDIA 提供的 PyTorch 容器和 AMP 在超参数优化过程中实现快速训练。该方法通过 5 倍交叉验证进行训练,并从每个折叠中保留表现最佳的检查点。总的来说,保存了 25 个模型检查点,但使用自适应加密,只有一半用于最终预测。由于该方法是完全基于 CNN 的,推理时间很快,并且可以在整个输入图像上一步完成,无需任何滑动窗口。单模型推理只需不到一秒钟的时间,这使得高通量和几乎实时的结果成为可能,这在临床环境中非常重要。
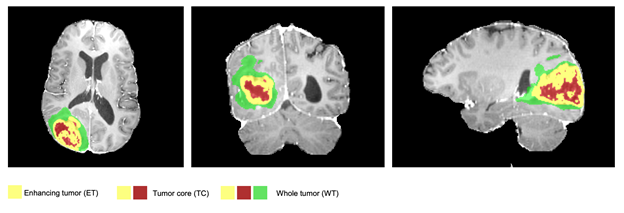
图 2 。一个典型的分割示例,预测的标签覆盖在 MRI 轴向、矢状面和冠状面切片上。整个肿瘤( WT )类别包括所有
可见标签(绿色、黄色和红色标签的结合),肿瘤核心( TC )类别为
红色和黄色的结合,增强肿瘤核心( ET )级别显示为黄色
(过度活跃的肿瘤部分)。
Swin UNETR :用于脑肿瘤三维语义分割的移位窗口 transformers –排名第 7
在 BraTS 挑战赛中排名第七的 Swin UNETR 是一个基于 transformer 的模型,而不是 CNN 模型。在 MONAI 中实现,它在整个肿瘤、肿瘤核心和增强肿瘤分割类中的平均 Dice 得分为 92 。 94% , Ha USD orff 距离为 1 。 7 。
transformers 是一类新的基于深度学习的模型,用于序列到序列的预测任务。在其原始公式中,它们由编码器和解码器组成。编码器由多个层次组成,多个层次的感知器跟随多个层次的自我注意。每个模块的输入通过剩余块添加到输出,并进行归一化。自我注意层学习从隐藏层计算的值的加权和,可以突出给定输入序列的重要特征。虽然它们最初被提议用于自然语言处理中的机器翻译任务,但后来它们已成功应用于计算机视觉和蛋白质药物生成等其他领域,并在各种基准测试中取得了最先进的性能。在计算机视觉中, transformer s 在各种基准方面取得了新的最先进的表现。 Swin UNETR 是一种利用 GPU 体系结构和性能不断进步来构建模型的新方法。
Swin transformers 是分层 transformer s ,其表示是使用移位窗口( Swin )计算的。这些 transformer 非常适合计算机视觉任务,如目标检测、图像分类、语义分割等。 Swin transformers 可以更有效地模拟两个域之间的差异,例如对象比例的变化和图像中像素的高分辨率,并且可以作为通用的视觉管道。该 NVIDIA SWN UNETR 模型利用 SWN transformer 编码器直接利用输入数据的 3D 补丁,而不依赖 CNN 进行特征提取。这使得 Swin UNETR 能够访问输入数据中的上下文多模态信息,并将其作为标记化嵌入 transformer 编码器进行有效处理。然后,基于 transformer 的 Swin UNETR 编码器通过跳过连接连接到 U 形架构中的 CNN 解码器,以进行最终分段预测。
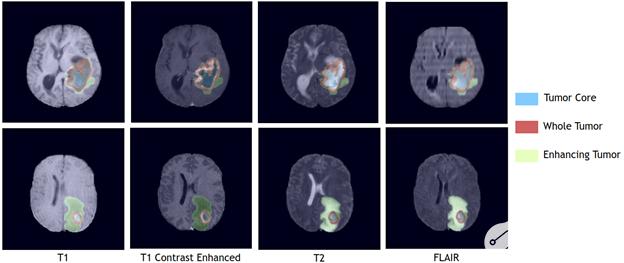
图 3 。使用 BraTS2021 验证数据集,覆盖在 4 种单独成像模式上的 TC 、 WT 和 ET 区域的 Swin UNETR 分割输出。
NVIDIA 的 SWI UNETR 模型在 NVIDIA DGX-1 团簇上使用八个 GPU 进行训练,初始学习率为 0 。 0008 ,并使用 AdamW 优化算法。使用 128 * 128 * 128 输入数据的随机面片以及随机轴镜像翻转和强度偏移的数据增强策略。 每轮培训需要 24 小时才能完成。与常用的基于 CNN 的分割模型相比, Swin UNETR 在失败次数方面更有效,在可训练参数数量方面具有中等的模型复杂度。它可以被有效地训练和用于推理。对于模型优化,使用一个通用的软骰子损失函数学习分割不同的脑肿瘤区域,每个类有一个单独的输出通道。在整个 BRATS21 训练集上使用 5 倍交叉验证方案对模型进行训练,其中通过平均 2 个不同 5 倍交叉验证的 10 个模型的输出来计算最终分割输出。
从 MONAI 开始。
在这里查找 BRAT 参与者的完整排行榜。

关于作者
Vanessa Braunstein 在 NVIDIA 的医疗团队从事产品营销工作。此前,她在基因组学、医学成像、制药、化学和诊断公司从事产品开发和营销。她学习分子和细胞生物学、公共卫生和商业。
审核编辑:郭婷
-
ARM处理器中SWI异常中断处理程序的实现2017-01-10 5782
-
NVIDIA 在首个AI推理基准测试中大放异彩2019-11-08 4748
-
Nvidia GPU风扇和电源显示ERR怎么解决2021-12-30 2297
-
在Ubuntu上使用Nvidia GPU训练模型2022-01-03 2414
-
移植中底层接口里的__swi关键字在SDT中能用吗2022-11-10 1370
-
swi中断程序是如何获得烧到RAM中系统函数的地址?2023-02-16 1528
-
在SWI软中断指令中LR中放的是异常模式下的返回地址求解2023-02-27 1115
-
NVIDIA Clara医疗成像AI模型在MD.ai项目中的应用2022-04-15 3802
-
新型transformer模型实现3D医学图像分析最新基准2022-08-15 7315
-
NVIDIA 人工智能开讲 | 回顾近期 NVIDIA 在医疗与生命健康领域的重点发布2023-05-15 1049
-
NVIDIA Ampere 架构的结构化稀疏功能及其在搜索引擎中的应用2023-07-18 1423
-
AI大模型在医疗领域起飞2023-07-25 2774
-
NVIDIA 知乎精彩问答甄选 | 发掘 NVIDIA 医疗行业相关精彩问答2023-09-06 1071
-
NVIDIA加速计算如何推动医疗健康2024-11-20 965
-
如何本地部署NVIDIA Cosmos Reason-1-7B模型2025-07-09 624
全部0条评论

快来发表一下你的评论吧 !

