

如何使用医学三维图像分割笔记本在MRI图像中预测脑肿瘤
描述
图像分割通过将数字图像的表示形式转换为更有意义、更易于分析的内容,将数字图像分割成多个部分。在医学成像领域,图像分割可以帮助识别器官和异常,测量它们,分类它们,甚至发现诊断信息。它通过使用从 x 射线、磁共振成像( MRI )、计算机断层扫描( CT )、正电子发射断层扫描( PET )和其他格式收集的数据来实现这一点。
为了实现能够为用例提供所需精度和性能的最新模型,您必须设置正确的环境,使用理想的超参数进行训练,并对其进行优化以达到所需的精度。所有这些都可能很耗时。数据科学家和开发人员需要一套合适的工具来快速克服繁琐的任务。这就是我们建立 NGC 目录的原因。
NGC 目录 是 GPU 优化 AI 和 HPC 应用程序和工具的集线器。 NGC 提供了对性能优化容器的方便访问,通过预训练模型缩短了模型开发时间,并提供了特定于行业的 SDK 来帮助构建完整的 AI 解决方案和加快 AI 工作流。这些不同的资产可以用于各种用例,从计算机视觉和语音识别到语言理解。潜在的解决方案涵盖汽车、医疗保健、制造和零售等行业。
图 1 NGC 目录, GPU 优化人工智能软件的集线器
基于 U-Net 的三维医学图像分割
在这篇文章中,我们将展示如何使用 医学三维图像分割笔记本 在 MRI 图像中预测脑肿瘤。这个职位是适合任何人谁是新的人工智能和有一个特别的兴趣在图像分割,因为它适用于医学成像。 3D-U-Net 实现了三维体的无缝分割,具有较高的精度和性能。它可以用来解决许多不同的分割问题。
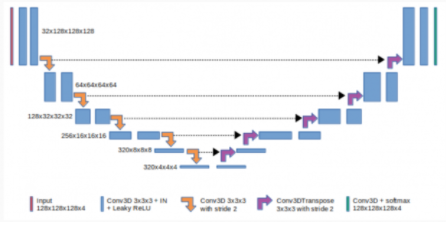
图 2 U-Net 模型概述
图 2 显示了 3du-Net 由收缩(左)和扩张(右)路径组成。它重复应用未添加的卷积,然后使用最大池进行下采样。
在深度学习中,卷积神经网络( CNN )是深度神经网络的一个子集,主要用于图像识别和图像处理。 CNN 使用深度学习来执行生成性和描述性任务,通常使用机器视觉以及推荐系统和自然语言处理。
CNNs 中的 Padding 指的是 CNN 内核处理图像时添加到图像中的像素数。未添加的 CNNs 意味着没有像素添加到图像中。
合用是 CNN 的一种下采样方法。最大池是一种常见的池方法,它总结了功能最活跃的存在。扩展路径中的每一步都包括特征映射的上采样和与压缩路径中相应裁剪的特征映射的连接。
Requirements
此资源包含一个 Dockerfile ,它扩展了 TensorFlow NGC 容器并封装了一些依赖项。可以使用以下命令下载资源:
wget --content-disposition https://api.ngc.nvidia.com/v2/resources/nvidia/unet3d_medical_for_tensorflow/versions/20.06.0/zip -O unet3d_medical_for_tensorflow_20.06.0.zip除了这些依赖项之外,还需要以下组件:
NVIDIA 码头工人
NGC 最新 TensorFlow 集装箱
NVIDIA 安培结构, NVIDIA 图灵,或 NVIDIA 伏特 GPU
要使用具有张量核心的混合或 TF32 精度或使用 FP32 来训练模型,请使用脑肿瘤分割数据集上 3D U-Net 模型的默认参数执行以下步骤。
下载资源
通过单击 资源页 右上角的三个点手动下载资源。
图 3 NGC 目录上 3D U-Net 资源的资源登录页
也可以使用以下 wget 命令:
wget --content-disposition https://api.ngc.nvidia.com/v2/resources/nvidia/unet3d_medical_for_tensorflow/versions/20.06.0/zip -O unet3d_medical_for_tensorflow_20.06.0.zip
构建 U-Net TensorFlow NGC 容器
此命令使用 Dockerfile 创建一个名为 unet3d_tf 的 Docker 映像,自动下载所有必需的组件。
docker build -t unet3d_tf 。
下载数据集
数据可在 脑肿瘤分割数据集 网站注册获得。应下载数据并将其放置在容器中安装 /data 的位置。
运行容器
要在 NGC 容器中启动交互式会话以运行预处理、训练和推断,必须运行以下命令。这将启动容器并将 。/data 目录作为卷装载到容器中的 /data 目录,将 。/results 目录装载到容器中的 /results 目录。
使用容器的优点是它将所有必需的库和依赖项打包到一个单独的、隔离的环境中。这样您就不必担心复杂的安装过程。
mkdir data
mkdir results
docker run --runtime=nvidia -it --shm-size=1g --ulimit memlock=-1 --ulimit stack=67108864 --rm --ipc=host -v ${PWD}/data:/data -v ${PWD}/results:/results -p 8888:8888 unet3d_tf:latest /bin/bash
启动容器内的笔记本
使用此命令在容器内启动 Jupyter 笔记本:
jupyter notebook --ip 0.0.0.0 --port 8888 --allow-root
使用以下命令将数据集移动到容器内的/ data 目录。 下载笔记本 :
wget --content-disposition https://api.ngc.nvidia.com/v2/resources/nvidia/med_3dunet/versions/1/zip -O med_3dunet_1.zip
然后,将下载的笔记本上传到 JupyterLab 中,运行笔记本的单元格对数据集进行预处理,并对模型进行训练、基准测试和测试。
Jupyter 笔记本
通过运行这个 Jupyter 笔记本的细胞,你可以首先检查下载的数据集并看到脑肿瘤图像。然后,查看数据预处理命令,准备数据进行训练。下一步是训练模型,并使用训练过程中的检查点作为预测步骤。最后,直观地检查预测函数的输出。
检查图像
要检查数据集,可以使用 nibabel ,这是一个提供对一些常见的医学和神经成像文件格式的读/写访问的包。
通过运行接下来的三个单元格,您可以使用 pip install 安装 nibabel ,从数据集中选择一个映像,并使用 matplotlib 从数据集中打印所选的第三个映像。您可以通过更改代码中的图像地址来检查其他数据集图像。
import nibabel as nib
import matplotlib.pyplot as plt
img_arr = nib.load('/data/MICCAI_BraTS_2019_Data_Training/HGG/BraTS19_2013_10_1/BraTS19_2013_10_1_flair.nii.gz').get_data()
def show_plane(ax, plane, cmap="gray", title=None):
ax.imshow(plane, cmap=cmap)
ax.axis("off")
if title:
ax.set_title(title)
(n_plane, n_row, n_col) = img_arr.shape
_, (a, b, c) = plt.subplots(ncols=3, figsize=(15, 5))
show_plane(a, img_arr[n_plane // 2], title=f'Plane = {n_plane // 2}')
show_plane(b, img_arr[:, n_row // 2, :], title=f'Row = {n_row // 2}')
show_plane(c, img_arr[:, :, n_col // 2], title=f'Column = {n_col // 2}')
结果如图 4 所示。
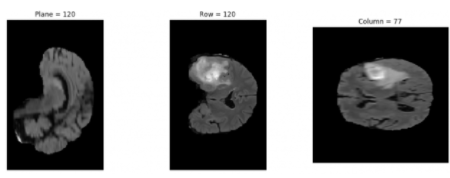 图 4 .作为 Jupyter 笔记本的一部分包含在数据集中的图像的示例输出
图 4 .作为 Jupyter 笔记本的一部分包含在数据集中的图像的示例输出数据预处理
dataset/preprocess_data.py 脚本将原始数据转换为用于培训和评估的 TFRecord 格式。该数据集来自 2019 年布拉特挑战赛 ,包含超过 3 TB 的多机构、常规、临床获得、术前、多模式、胶质母细胞瘤( GBM / HGG )和低级别胶质瘤( LGG )的 MRI 扫描,并经病理证实诊断。如果可用,还包括患者的总体生存率( OS )数据。这些数据是在训练、验证和测试数据集中构建的。
图像的格式是 nii.gz. NIfTI 是神经成像的一种文件格式。可以通过运行以下命令对下载的数据集进行预处理:
python dataset/preprocess_data.py -i /data/MICCAI_BraTS_2019_Data_Training -o /data/preprocessed -v
处理后的图像的最终格式是 tfrecord 。为了帮助您高效地读取数据,请序列化数据并将其存储在一组文件中(每个文件大约 100 到 200 MB ),每个文件都可以线性读取。如果数据是通过网络传输的,这一点尤其正确。它还可以用于缓存任何数据预处理。 TFRecord 格式是一种用于存储二进制记录序列的简单格式,它大大加快了数据加载过程。
使用默认参数进行培训
启动 Docker 容器后,可以使用默认的超参数(例如,{ 1 到 8 } GPU s { TF-AMP / FP32 / TF32 })开始单个折叠(折叠 0 )的训练:
Bash examples/unet3d_train_single{_TF-AMP}.sh
例如,要以 32 位精度( FP32 或 TF32 )在一个 GPU 上以批大小 2 运行,请运行以下命令:
bash examples/unet3d_train_single.sh 1 /data/preprocessed /results 2
要训练具有混合精度( TF-AMP )的单折叠,每个 GPU 有八个 GPU 个,每 GPU 批大小为 2 ,请运行以下命令:
bash examples/unet3d_train_single_TF-AMP.sh 8 /data/preprocessed /results 2
培训绩效基准
可以通过运行基准脚本来评估培训绩效:
bash examples/unet3d_{train,infer}_benchmark{_TF-AMP}.sh
此脚本使模型运行并报告性能。例如,要在四个 GPU 上使用批量为 2 的 TF-AMP 对培训进行基准测试,请运行以下命令:
bash examples/unet3d_train_benchmark_TF-AMP.sh 4 /data/preprocessed /results 2
Predict
您可以使用测试数据集和 predict as exec 模式来测试模型。结果保存在 model_dir 目录中, data_dir 是数据集的路径:
python main.py --model_dir /results --exec_mode predict --data_dir /data/preprocessed_test
绘制预测结果
在下面的代码示例中,您将从 results 文件夹打印所选结果之一:
import numpy as np
from mpl_toolkits import mplot3d
import matplotlib.pyplot as plt
data= np.load('/results/vol_0.npy')
def show_plane(ax, plane, cmap="gray", title=None):
ax.imshow(plane, cmap=cmap)
ax.axis("off")
if title:
ax.set_title(title)
(n_plane, n_row, n_col) = data.shape
_, (a, b, c) = plt.subplots(ncols=3, figsize=(15, 5))
show_plane(a, data[n_plane // 2], title=f'Plane = {n_plane // 2}')
show_plane(b, data[:, n_row // 2, :], title=f'Row = {n_row // 2}')
show_plane(c, data[:, :, n_col // 2], title=f'Column = {n_col // 2}')
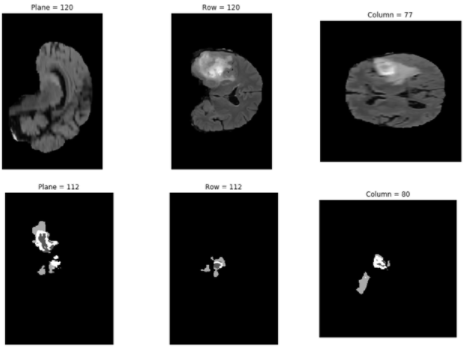 图 5 .模型预测的结果与图 4 所示的实际图像相对应。
图 5 .模型预测的结果与图 4 所示的实际图像相对应。高级选项
对于那些希望探索此笔记本内置高级功能的用户,可以使用 -h 或 --help 查看 main 。 py 可用选项的完整列表。通过运行下一个单元格,您可以看到如何更改此脚本的执行模式和其他参数。使用此脚本,可以使用自定义的超参数执行模型训练、预测、评估和推断。
python main.py --help
main.py 参数可以更改以执行不同的任务,包括训练、评估和预测。
也可以使用默认超参数训练模型。通过运行 python main.py --help 命令,可以看到可以更改的参数列表,包括训练超参数。例如,在训练模式下,可以使用以下命令将学习速率从默认的 0 。 0002 更改为 0 。 001 ,并将训练步骤从 16000 更改为 1000 :
python main.py --model_dir /results --exec_mode train --data_dir /data/preprocessed_test --learning_rate 0.001 --max_steps 1000
您可以运行 main.py 中提供的其他执行模式。例如,在本文中,我们通过运行以下命令使用了 python.py 的预测执行模式:
python main.py --model_dir /results --exec_mode predict --data_dir /data/preprocessed_test
总结和下一步
在这篇文章中,我们展示了如何使用一个简单的 NGC 目录中的 Jupyter 笔记本 开始使用医学成像模型。当您从这个 Jupyter 笔记本转换到构建您自己的医学成像工作流时,考虑使用 NVIDIA Clara 列。 Clara 列车 包括人工智能辅助注释 API 和注释服务器,可以无缝地集成到任何医疗查看器中,使其具有 AI 能力。培训框架包括分散式学习技术,例如针对 AI 工作流的联合学习和转移学习。
关于作者
Shokoufeh Monejzi Kouchak 是 NVIDIA 的技术营销工程师,专注于深度学习模型。肖库菲从亚利桑那州国家大学获得了计算机工程学博士学位,她把重点放在驾驶行为分析和驾驶员注意力检测上,并用深度学习模型。
审核编辑:郭婷
-
比较好的三维图像绘制的函数有哪些啊2011-09-30 0
-
labview 三维图像显示不出来,求助~2014-04-08 0
-
labview前面板网页发布后,浏览器为何无法显示三维图片控件的图像?2017-04-18 0
-
基于FPGA+DSP的三维图像信息处理系统设计2019-06-24 0
-
深度学习在医学图像分割与病变识别中的应用实战2023-09-04 0
-
基于激光差频扫描的水下三维图像2009-12-14 943
-
三维图像信息处理FPGA+DSP核心架构的设计与实现2009-01-09 1035
-
三维图像详解2009-08-03 2724
-
基于FPGA的医学图像三维重建系统设计与实现2011-03-15 1113
-
以FPGA+DSP为核心架构的实时三维图像信息处理系统设计2018-07-19 1483
-
使用NI LabVIEW设计了三维图像设计2018-06-22 7557
-
点云的概念以及与三维图像的关系2021-08-17 7343
-
使用深度学习进行三维图像处理2021-11-05 3307
-
一文详解点云及三维图像处理技术2023-03-31 3102
-
20个MATLAB三维图像绘制的示例演示2023-07-07 2839
全部0条评论

快来发表一下你的评论吧 !

