

CellTrek单细胞空间转录组联合分析
描述
自单细胞测序技术横空出世以来,由于其可以将测序缩小到单个细胞的单位内进行,对于更加全面精确地了解生物基因表达情况具有重要意义。但是,由于单细胞测序本身的技术特点,尽管我们可以得到单个细胞的表达信息,但是细胞在原本组织内的空间位置信息是完全丢失的,这些信息在理解细胞微环境或细胞间互作时同样关键。
为了解决这个问题,空间转录组技术出现了,这项技术可以在组织原位上以接近单个细胞大小的尺度来进行转录组测序,但因为技术限制以及生物组织本身的复杂性,空间转录组还不能精确提供单个细胞尺度上的转录组信息。
单细胞测序技术具有单个细胞的精度,空间转录组提供了组织原位的信息,因此将两者结合起来就能近似得到完整的信息。目前已经有许多分析工具试图将空间信息与单细胞信息整合起来,并且取得了一定的进展。本文将介绍的CellTrek就是其中之一。
目前常用的整合算法有许多,例如去卷积,通过推断spot内的细胞组成来将两者整合在一起。而CellTrek则另辟蹊径,通过直接推断单细胞的空间坐标来实现信息的补全。Cell Trek的计算原理可见下图。简单来说,该方法首先将单细胞数据与空间转录组数据整合,共嵌入到(coembed)共同的隐空间内,然后从中抽出空间转录组的数据作为训练数据集,使用随机森林模型(Random Forests,RF)从spot的基因表达情况预测其空间坐标。这里所依据的基本思路就是,细胞在组织中的分布并不是随机混乱的,同一类细胞大多都是集中于同一片区域。
空间转录组的数据还会同时进行非线性插值增强其空间分辨率。然后,将整个共嵌入矩阵输入训练好的模型,生成一个衡量单细胞与空间spot之间表达相似性的RF距离矩阵,基于这个矩阵使用相互最近邻域算法(mutual nearest neighbors,MNN)生成一个spot-细胞稀疏图,最后根据细胞在稀疏图上的相邻spot推断出细胞的空间坐标。因为相同类型的细胞拥有较为相似的基因表达,所以基于空间转录组数据训练的模型可以用来预测细胞的位置坐标。
01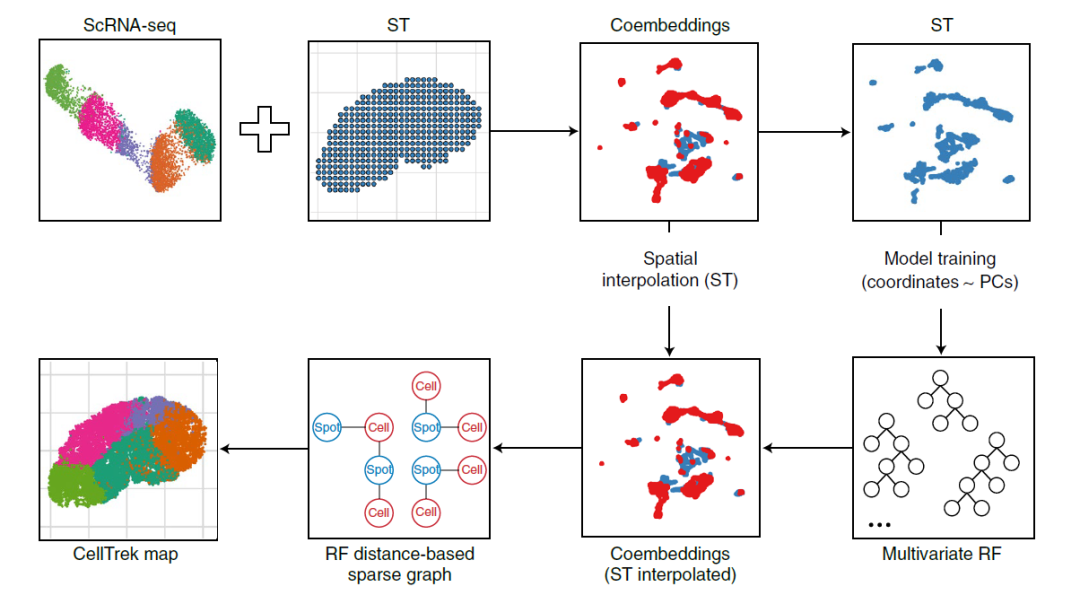
这一算法的优势在于将细胞直接定位到组织切片照片上,而不是像去卷积类方法那样计算每个spot内的细胞类型比例。直接定位带来的另一个好处就是在接下来的数据分析中可操作性大大提高了,像去卷积类方法得到的细胞比例矩阵我们很难再对其进行什么操作,但是我们可以对CellTrek 整合后的结果进行细胞共定位分析、基因空间权重共表达分析等等,因为他本质仍然是单细胞数据,只是附带上了空间坐标。
以小鼠大脑皮层的空转数据与单细胞数据为例,我们最后得到的结果会是这样的。可以看到不同类型的细胞在空间上呈现出明显的层状分布,这符合大脑皮层的结构特征,并且不同的细胞类型出现在了它们应该出现的皮层位置。 02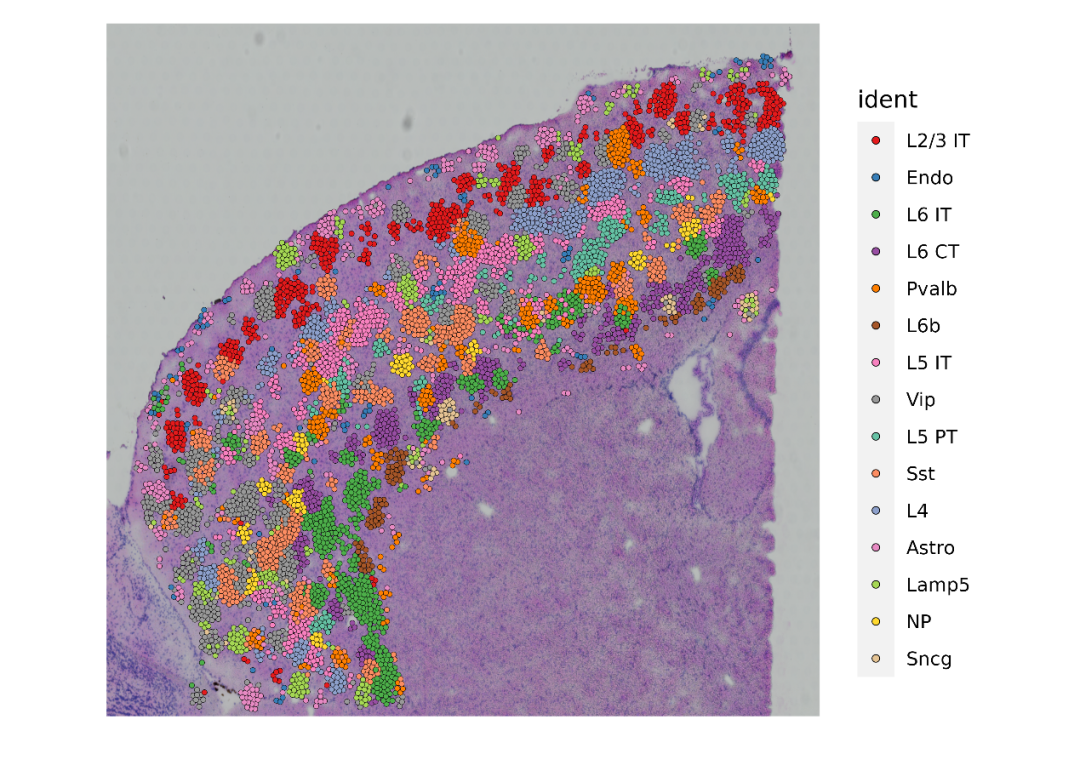
内测数据结果
CellTrek的缺点在于,第一,对计算资源的需求比较大,当单细胞数据量较大时会花费比较长的时间;第二,对于空转数据与单细胞数据的匹配度要求比较高,也就是说空转数据与单细胞数据最好取自同一块组织区域,不要出现无法匹配的空间区域或细胞类型,否则最终结果会非常混乱,如下图。
03
内测数据结果
总而言之,CellTrek提供了一条不同于当下其他单细胞-空转整合分析方法的道路,结果更加接近生物组织中的实际情况,并为整合后的下游分析提供了便利。
审核编辑:刘清
-
什么是高通量单细胞RNA测序技术?2019-04-25 10897
-
厦门大学研发出全新高通量单细胞转录组测序方法2020-06-02 4299
-
单细胞转录组学技术的十年2020-09-23 4523
-
微流控芯片技术在单细胞基因组学研究中的应用2022-03-03 2901
-
空间多组学测序技术的意义、原理及实验流程2022-05-16 5394
-
mTORC1高活性的干细胞样状态肿瘤细胞类型分析2022-10-10 2876
-
基于单细胞与空间转录组数据评估细胞间互作分析算法2022-11-03 1995
-
基于微流控平台的单细胞通讯研究进展2022-11-29 1686
-
单细胞+转录组测序:揭示GABA信号调控神经发生2023-01-12 1549
-
德运康瑞完成近亿元A+轮融资,领跑单细胞空间组学临床应用2023-02-08 1680
-
聚类分析中的机器学习与统计方法综述(一)2023-05-19 1819
-
使用 RAPIDS 进行更快的单细胞分析2023-07-05 1371
-
基于微流控的低成本空间转录组测序方法2023-08-24 2071
-
基于精准聚焦目标空间区域的转录组和蛋白质组一站式解决方案2023-08-29 1858
-
Visium空间转录组和单细胞转录组的区别2023-08-30 5842
全部0条评论

快来发表一下你的评论吧 !

