

基于微流控的低成本空间转录组测序方法
描述
空间转录组学技术是揭示组织精细解剖位置的组学工具。基于高通量测序的空间转录组学技术具有检测结果无偏移、使用门槛低等优势。目前,科学家开发了多种技术构建条形码RNA捕获阵列,然而难以实现成本、探针编码复杂度、基因捕获灵敏度和空间分辨率之间的平衡关系。
近日,吉林大学张俊虎、梁重阳团队在预印本bioRxiv上发表了题为“Well-ST-seq: cost-effective spatial transcriptomics at cellular level and high RNA capture efficiency”的文章,报道了一种低成本、细胞分辨率水平、高RNA捕获效率的空间转录组测序(ST-seq)方法。
该技术将带有高密度活性基团的微珠排列于微井阵列,再采用微流控技术对微珠阵列进行编码,生成高密度的RNA捕获探针,其灵敏度可达3896 UMIs/10微米。该探针阵列制备全程未用到大型精密仪器,具有小型化、低成本优势,构建成本约为1.21美元/mm²。
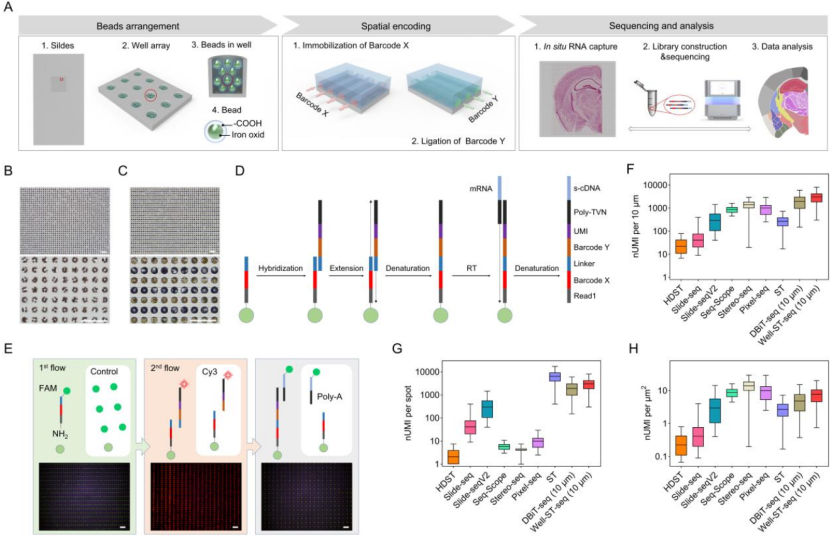
Well-ST-seq技术概述
此外,该技术空间分辨率由微孔道及微井阵列尺寸控制,可实现10微米分辨率的空间转录组测序。
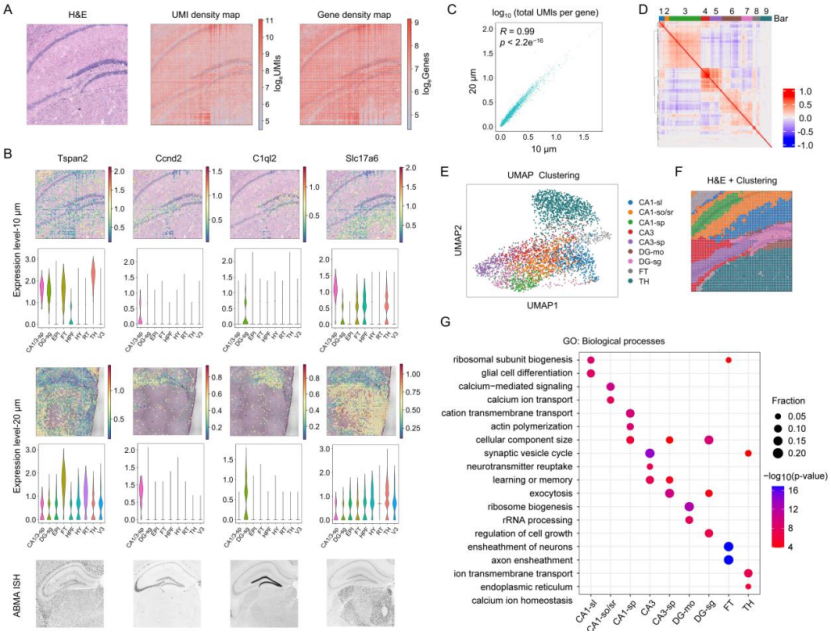
通过Well-ST-seq-10 μm绘制小鼠海马的高空间分辨率图谱
为了验证Well-ST-seq在高分辨率空间转录组测序方面的灵活性,研究团队制作了10 μm分辨率的微通道和微孔阵列,并选择小鼠大脑的海马区域进行组织切片。Well-ST-10 μm和Well-ST-20 μm数据中标记基因的空间表达图谱显示出了高度的一致性。Well-ST-10 μm检测到的标记基因表达与Allen小鼠脑图谱(AMBA)的ISH数据一致,进一步验证了Well-ST-seq的准确性。
而与Well-ST-seq-20 μm数据相比,Well-ST-seq-10 μm生成的空间地图显示出更详细的解剖形态,与组织切片的HE染色图像一致;与基于ABMA注释结果的解剖学定义的脑区很好地对应,并且集群的标记基因表现出差异表达模式。结果表明,Well-ST-seq能够进行高分辨率的空间转录组学分析。
总而言之,该技术为空间单细胞转录组测序提供了低成本的解决方案,且实现了高灵敏度、分辨率、可伸缩性以及与现有数据库的兼容性,是一种高效、经济、便利的原位捕获和分析方法,为空间转录组学研究开辟了新的可能性。
审核编辑:刘清
-
全基因组测序的优势 精选资料分享2021-07-29 1611
-
全基因组测序的优势是什么?2021-10-27 1514
-
最新型的低成本、便携设备助力单细胞测序临床应用已被开发出来2019-05-20 1244
-
厦门大学研发出全新高通量单细胞转录组测序方法2020-06-02 4296
-
单细胞转录组学技术的十年2020-09-23 4521
-
空间转录组研究将对基因测序领域带来的三大影响及作用2020-11-10 6678
-
空间转录组测序用于免疫治疗研究2020-11-18 3429
-
空间多组学测序技术的意义、原理及实验流程2022-05-16 5386
-
单细胞+转录组测序:揭示GABA信号调控神经发生2023-01-12 1548
-
新型定量微流控装置用于促进轴突转录组分析2023-06-02 1702
-
Visium空间转录组和单细胞转录组的区别2023-08-30 5839
-
基于液滴微流控的单细胞测序法可提升mRNA捕获效率2023-09-15 2829
-
高通量测序技术及原理介绍2024-02-03 17774
-
低成本微流控芯片的加工与键合方法2024-07-23 2209
全部0条评论

快来发表一下你的评论吧 !

