

一种基于液滴微流控的高通量单细菌细胞RNA测序法介绍
描述
单细胞RNA测序(scRNA-seq)方法自2005年创立以来,已广泛应用于真核生物领域。目前流行的基于液滴的scRNA-seq系统(如10× Genomics Chromium平台)的关键策略是利用液滴微流控技术将单细胞与唯一的DNA条形编码珠子共同封装在液滴中,随后由珠子上数十亿份唯一的条形编码poly(T)引物捕获多聚腺苷酸化的mRNA。
然而,这种策略与细菌的单细胞RNA测序并不兼容。其中一个主要原因是细菌mRNA缺乏3′端poly(A)尾部,而这正是poly(T)引物的捕获位点。此外,典型细菌的RNA含量比典型哺乳动物细胞低两个数量级,核糖体RNA(rRNA)占细菌总RNA的80%以上,这使得在单个细菌细胞水平上有效捕获和区分这些低含量mRNA变得十分困难,坚韧的细胞壁也使细菌难以裂解并释放液滴中的RNA。
为了解决这些问题,人们开发了一些相对低通量的单细菌RNA测序方法,这些方法需要使用单细胞操纵器或荧光激活细胞分选术(FACS)将单细菌分离到多孔板中。之前开发的两种细菌RNA测序方法“PETRI-seq”和 “microSPLiT”使用随机反转录(RT)引物,使研究人员能够通过分割池条形码策略同时分析数千个固定细菌,这极大地增强了大规模研究细菌群落转录组异质性的能力。然而,这种组合式分裂池条形码策略需要在96孔板中进行多次反应,而96孔板并不是高通量单细胞RNA测序的常用或灵敏平台。此外,即使富集了mRNA,PETRI-seq和microSPLiT中的大部分映射读数也是rRNA,这导致了相当高的测序成本。
为了解决上述问题,近期,浙江大学王永成研究员课题组在Nature Communications杂志上发表了题为“Droplet-based high-throughput single microbe RNA sequencing by smRandom-seq”的研究论文,报告了一种基于液滴的高通量单细菌RNA测序方法“smRandom-seq”。
在这项工作中,研究人员利用随机引物原位生成互补DNA(cDNA),利用液滴微流控技术进行单细菌条形码编码,并利用基于CRISPR的rRNA去除法进行mRNA富集,在此基础上开发了一种高通量、高灵敏度的单细菌RNA测序方法“smRandom-seq”。
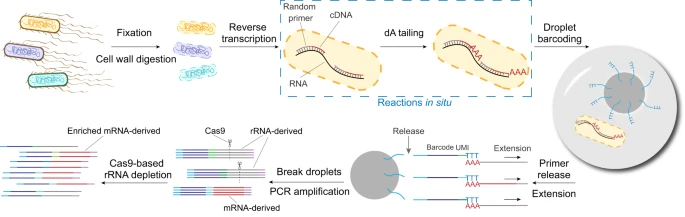
图1 基于液滴微流控的smRandom-seq测序方法
smRandom-seq具有条形码效率高、物种特异性高(99%)、交叉污染小(1.6%)和自动化能力强等特点。基于CRISPR的rRNA去除法显著降低了rRNA百分比(83%-32%)。由于高效的rRNA去除法,大肠杆菌的映射mRNA读数高度富集,增加了4倍(16%-63%)。在约8000个大肠杆菌群体中,每个细菌可检测到约1000个基因。研究人员还验证了smRandom-seq可用于其他常见细菌物种,包括革兰氏阴性菌(鲍曼不动杆菌、肺炎双球菌和铜绿假单胞菌)和革兰氏阳性菌(枯草杆菌和金黄色葡萄球菌)。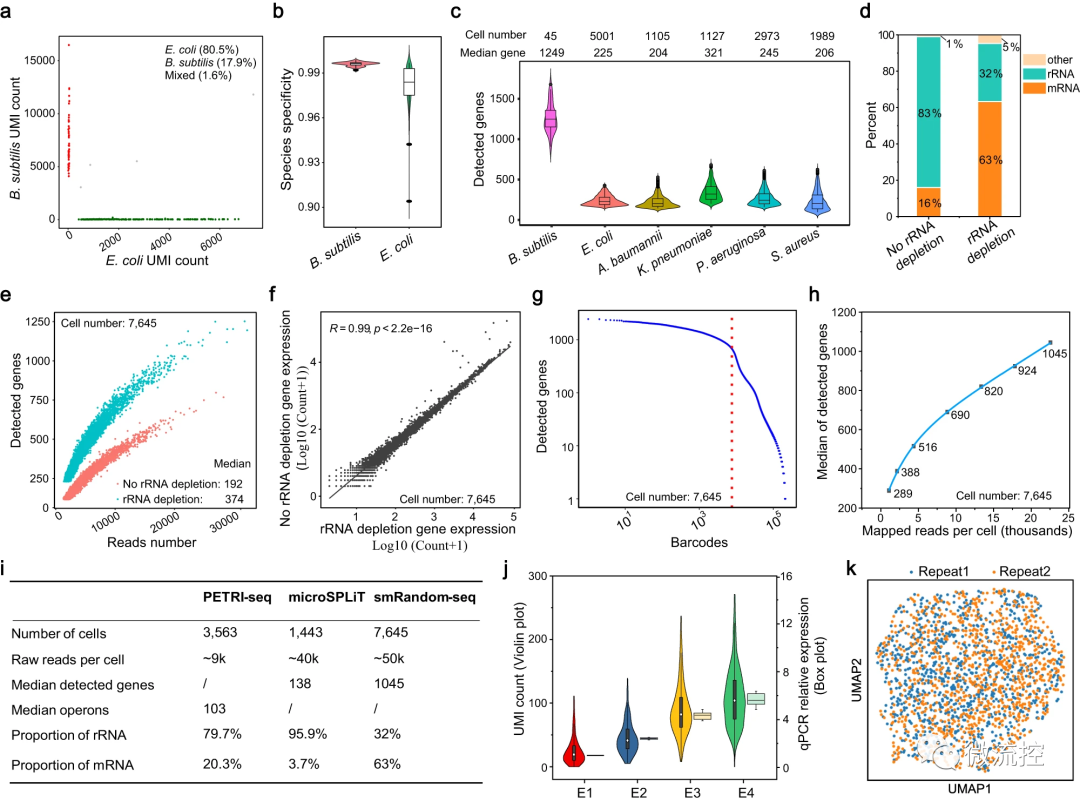
图2 使用参考细菌样本验证smRandom-seq测序方法的可行性
研究人员还发现大肠杆菌群在抗生素作用下表现出形态异质性,并进行 smRandom-seq以研究单个细菌的这些转录组变化,smRandom-seq对基因表达的聚类分析结果与形态特征一致。大肠杆菌的一些亚群显示出不同的SOS响应和代谢通路基因表达模式,这可能是抗生素耐药亚群。
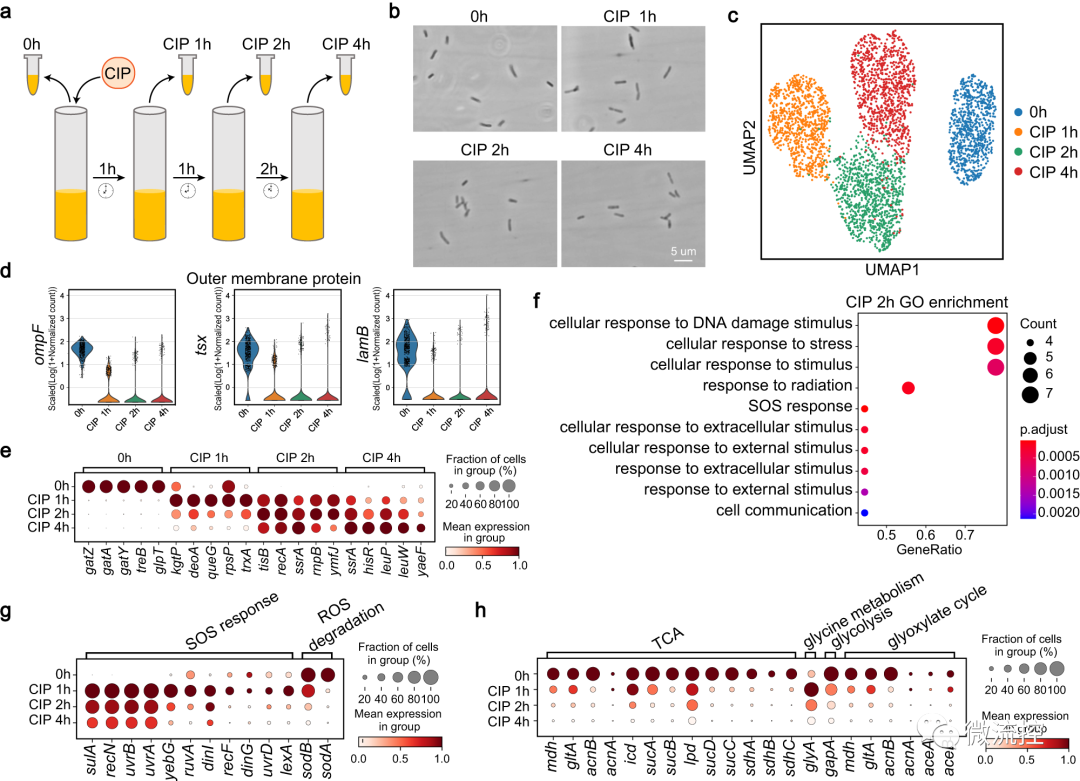
图3 利用smRandom-seq测序方法捕获单个大肠杆菌在抗生素胁迫下的转录组变化
总之,“smRandom-seq”测序方法提供了一种高通量单细菌转录组图谱分析工具,它将促进未来在细菌耐药性、持久性、细菌-宿主相互作用和微生物组研究方面的发现。
审核编辑:刘清
-
功率放大器在液滴微流控细胞分选中的应用2025-04-03 644
-
高通量测序技术及原理介绍2024-02-03 17791
-
基于液滴微流控的单细胞测序法可提升mRNA捕获效率2023-09-15 2832
-
用于高通量单液滴的操纵和生成的有源矩阵数字微流控芯片2023-08-14 3692
-
用于超高通量筛选的荧光偶联液滴微流控策略研究进展2023-06-05 1758
-
高通量测序数据分析:RNA-seq 精选资料分享2021-07-26 1448
-
10K Genomics是国内第一款液滴微流控单细胞测序仪2021-05-27 3413
-
厦门大学研发出全新高通量单细胞转录组测序方法2020-06-02 4300
-
基于旋转圆台的非微流控高通量微液滴生成装置2019-06-10 3184
-
什么是高通量单细胞RNA测序技术?2019-04-25 10901
-
高通量测序技术及其应用2018-02-28 16719
全部0条评论

快来发表一下你的评论吧 !

