

固态照明技术革新多路复用荧光检测
描述
长久以来,在复杂、异质的样品中同时识别以及定位多个分子或者分子组装的能力一直是推动荧光显微镜在生物和物理科学研究中应用的主要特性。例如,自1986年以来,使用四种光谱上的不同荧光团来识别DNA中的腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)和鸟嘌呤(G)碱基,这一直是大多数自动化DNA测序技术的基础。然而,对大规模生物系统的基因组和转录组的研究可能需要同时识别和定位成百上千的分子标靶。这种高度并行的分析超出了基于光谱鉴别的多路复用能力。Lumencor分析了基于光谱鉴别的多路复用荧光检测的局限性,以及为扩大可检测标靶数量而引入的一些固态光源新技术。
光谱鉴别的局限性
大多数多路复用检测方案都基于光谱鉴别,因为与基于时间或空间鉴别方法相比,它的技术复杂性较低,成本也较低。然而由于光谱串扰,光谱的鉴别方法范围仅限于大约5个目标(图1和图2)。这种局限性主要是由于用于双分子标记的荧光染料和荧光蛋白(FP)的光谱特性,如图1 所示。在此示例中,虽然只有两个荧光标记,但它们的激发和发射波长跨越了整个可见光波长范围(400-700nm),并且具有明显的光谱重叠,会导致光谱分离不完全。如果以485nm左右的光进行激发(灰色部分),两种荧光团会被同时激发。只有波长大于550nm时才能选择性地激发其中的一种,从而获得光谱鉴别。
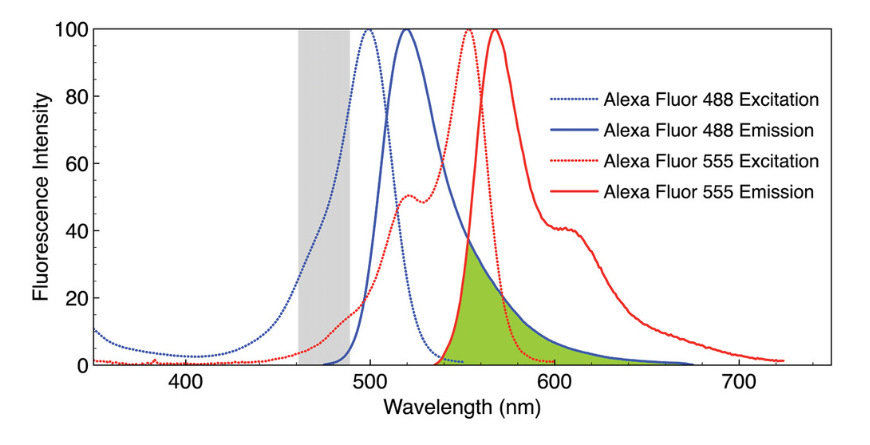
图1. Alexa Flour488和Alexa Fluor 555荧光染料的归一化荧光激发和发射光谱。发射光谱的重叠区域由绿色阴影表示。灰色阴影区域表示图2中用于采集图像A-C的激发带宽(475/28nm)。
针对串扰的问题,虽然已经开发出具有窄发射光谱的量子点纳米晶体,可以提供更好的分离光谱。但与有机染料相比,这种改进的代价是荧光团尺寸增加了一个数量级以上,这反过来又阻碍了它们在双分子标记应用中的应用。
Lumencor的固态光引擎优化了输出光谱,提供了多个窄线宽的光源,尽可能实现对特定荧光染料的精确激发,而这对于多重荧光检测至关重要,可以有效减小光谱串扰问题的发生。下图中是使用Lumencor SPECTRA光引擎、Andor Zyla 5.5 sCMOS相机和Nikon Ti2显微镜对麂皮成纤维细胞进行成像。可以看到由于激发光带宽(555/28 nm)与Alexa Fluor 488的激发光谱没有显著交叉,因此图像没有因串扰而退化。
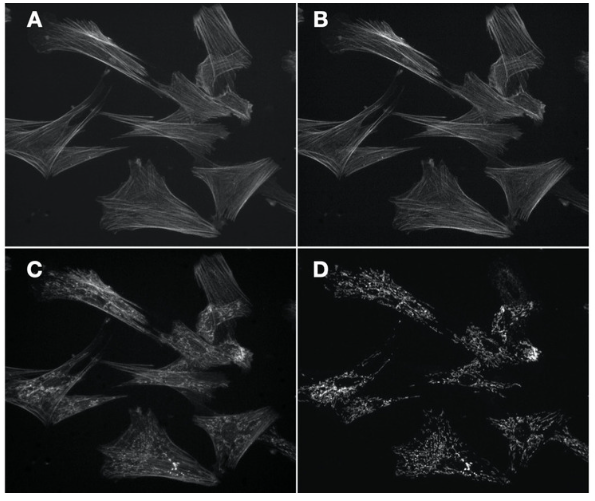
图2. 四张60倍荧光图像,展示了同一视野下的麂皮成纤维细胞。
用Alexa Fluor 488 phalloidin (actin)以及mouse anti–OxPhos Complex V + Alexa Fluor 555 goat anti–mouse IgG (mitochondria)进行标记。A.使用单波段激光和发散滤光片以及单波段的二色分光镜来获得的肌动蛋白细胞骨架的图像。B.使用单波段激发和发射滤光片以及多波段段二色分光镜来获得的等效肌动蛋白细胞骨架图像。C.由于光谱串扰导致线粒体检测时肌动蛋白细胞骨架图像退化。图像使用单波段激发滤光片、多波段二色分光镜和多波段的发射滤光片获得。D.使用单波段激发滤光片、多波段二色分光镜和多波段发射滤光片获得的线粒体图像。
多重荧光成像的突破
好马配好鞍,为了充分利用 Lumencor 光源的优秀性能,科研人员正在进一步开发和优化用于多路复用荧光检测的方案。
优化的光学滤光片和解混算法
来自美国国家神经系统疾病和卒中研究所的一篇Nature论文中有为解决多重荧光成像的荧光串扰问题提供了一些新策略。为了理解大脑中健康和病理生理过冲需要对多个组织样本进行大量的组织学观察,以表征所有主要的脑细胞类型。为了实现大规模多重免疫荧光成像技术,以实现多达10种不同荧光标记物的同时成像,从硬件上,他们优化了滤光片的组合,使用了10组自定义的激发/二向色/发射滤光片组合,这些精心挑选的滤光片被优化用于迭代成像不同的光谱兼容的荧光标记物,以确保在成像光谱范围内具有最小的光谱串扰。而从软件上,本文中也开发了多种算法以从复杂的脑组织样本中提取出丰富和准确的生物学信息,主要是一种半监督稀疏线性光谱解混算法,校正原始荧光图像数据的串扰和非特异性标记,提高了信号分离的准确性。

Lumencor固态光引擎SPECTRA的光谱输出针对本方案中使用的荧光染料面板的选择性激发进行了优化,如图3所示。光引擎集成了 LED、光管和激光器,以实现所需的光谱激发分布和所需的光功率。使用相同的荧光染料组合和滤光片配置,通过连续的剥离、重新标记和成像轮次,可以增加多重检测的程度。通过5个周期的剥离、10倍多重标记和成像,已证明可以在大鼠大脑冠状切片中同时定位50个生物标记靶标。
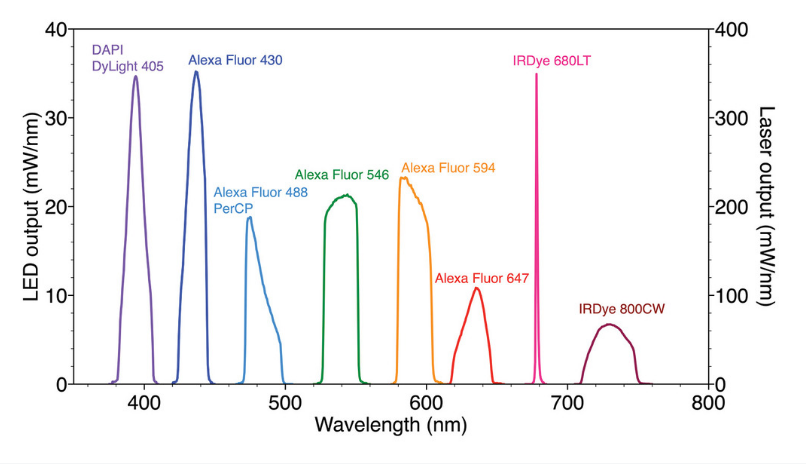
图3. SPECTRA光引擎的光谱输出经过优化,可选择性激发用于10倍免疫荧光检测的荧光染料。
参考文献:
D Maric, J Jahanipour, B Roysam et al., Whole-brain tissue mapping toolkit using large-scale highly multiplexed immunofluorescence imaging and deep neural networks. Nature Communications (2021)12:1550
MERFISH技术
通过在基于光谱鉴别的多路复用技术中添加空间和时间维度,已经开发出能够同时识别和定位数百和数千个生物分子标靶的多路复用技术。而用于基因表达分析的分子条形码扫描就是这样一种技术,利用核酸杂交来识别DNA和RNA分子标靶,并将其作为产生荧光染料顺序排列的模版。
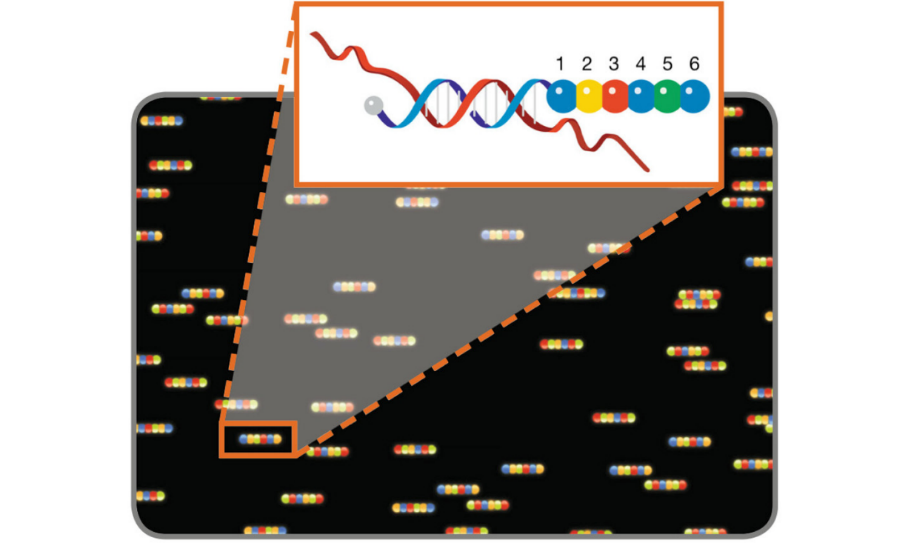
图4. 用于基因表达分析的分子条形码扫描。扫描仪配有激发和发射滤光片,可区分由蓝色、绿色、黄色和红色圆圈代表的 4 种染料,可读取探针位置 1 至 6 的荧光信号,以揭示杂交核酸靶标的编码身份。
以分子条形码扫描为出发点的MERFISH (多重误差鲁棒荧光原位杂交)是一种高度多重化的单分子成像技术,能够同时测量单个细胞中数百至数千个目标 RNA 种类的拷贝数和空间分布。首先将编码的寡核苷酸探针和目标RNA杂交,并依次添加荧光染料标记的读取核苷酸探针。这些读取探针会在单分子级别上被空间定位,然后被熄灭。读取探针必须仅与编码探针杂交,而不能与细胞核酸发生杂交。通过连续的成像周期积累编码探针的身份,如果编码探针通过读取探针的杂交被检测到,则赋予二进制位值“1”,如果没有检测到,则赋予“0”。原则上,需要 12 个杂交+成像周期来识别 4095(212-1)个独特的编码探针。实际上,二进制编码空间较少,以尽量减少假阴性(1>0)和假阳性(0>1)调用错误以及随之而来的靶标 RNA 错误识别。例如,已经证明可以使用 69 位条形码识别 10,050 个 RNA 靶标。
对于MERFISH应用,需要高强度、空间均匀的照明场,并且与典型sCMOS相机传感器的尺寸(~200mm2)相匹配。为了满足此要求,需要在显微镜中安装专业的临界照明器。Lumencor的CELESTA光引擎很好地满足了这些需求,通过光纤输出耦合到显微镜中,通过高数值孔径的高放大倍数物镜在样品平面上提供1-10W/mm2。
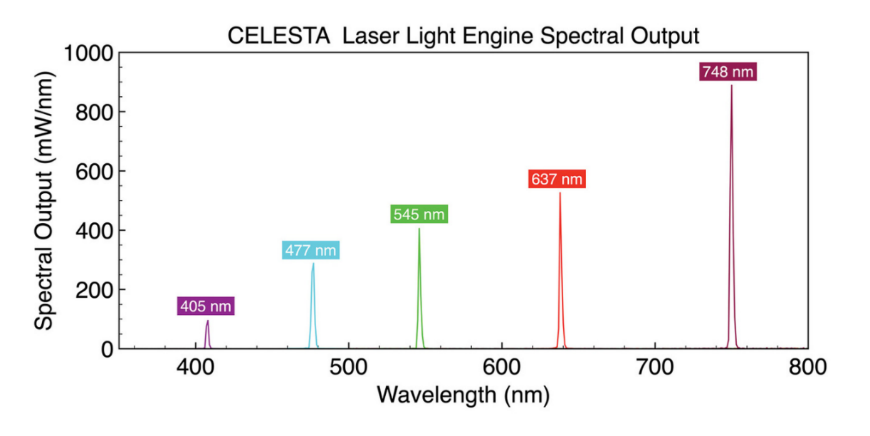
图5. 为MERFISH多路单分子成像优化的CELESTA激光光引擎输出光谱。
显然,对于那些超出简单光谱分辨能力的高并行荧光标记生物分析,可以通过精心选择荧光团、光学滤光片、精细调节激发照明和智能算法来实现。巧妙的标记策略,如分子条形码,也可以应用于广泛的生物样本,以增强来自高度复杂组织标本的生物分子信息的光谱分辨和空间分布。在此,我们讲述了一种大规模并行单分子成像技术,MERFISH,利用重复标记协议来最大化单细胞中数十万个RNA靶标的拷贝数量和空间分布信息。检索大量数据集的技术和机会对于当今丰富的基因组和转录组信息生物分析至关重要。实验者必须在有限的波长范围内进行光谱解析,以充分利用光谱宽度、光谱纯度和荧光团兼容性,以实现激发和发射之间的最佳区分。策略性的重复成像,辅以纯净、明亮、稳定的照明和高质量的光学玻璃,能够实现高度多重化的结果,并克服了在可用可见波长内定义的历史限制。
-
频分多路复用和时分多路复用的区别有哪些2024-05-07 6005
-
频分多路复用的原理 频分多路复用方式的分类2024-03-05 3082
-
如何在Mx1051的FlexCAN1中配置简单信号多路复用和扩展信号多路复用?2023-05-05 650
-
[6.4.1]--多路复用器李开鸿 2022-11-13
-
虹科案例|用于多路复用荧光检测的固态照明2022-07-21 2000
-
多路复用ICSP引脚如何控制开关?2020-04-01 2459
-
DWDM密波多路复用技术2010-05-12 718
-
什么是同步时分多路复用技术(STDM)2010-04-03 2743
-
时分多路复用(TDM),时分多路复用(TDM)的原理是什么?2010-03-19 11231
-
复用器的多路复用2010-01-07 1353
-
多路复用技术2009-06-27 1232
-
非多路复用与多路复用总线转换桥的设计与实现2009-03-28 1193
-
多路复用与数字复接2008-10-22 848
全部0条评论

快来发表一下你的评论吧 !

