

概率分布合成的数据上平均数的探索详细资料概述
电子说
描述
Philadelphia Media Network资深数据分析师Daniel McNichol使用R语言演示了毕达哥拉斯平均数在不同概率分布上的效果。
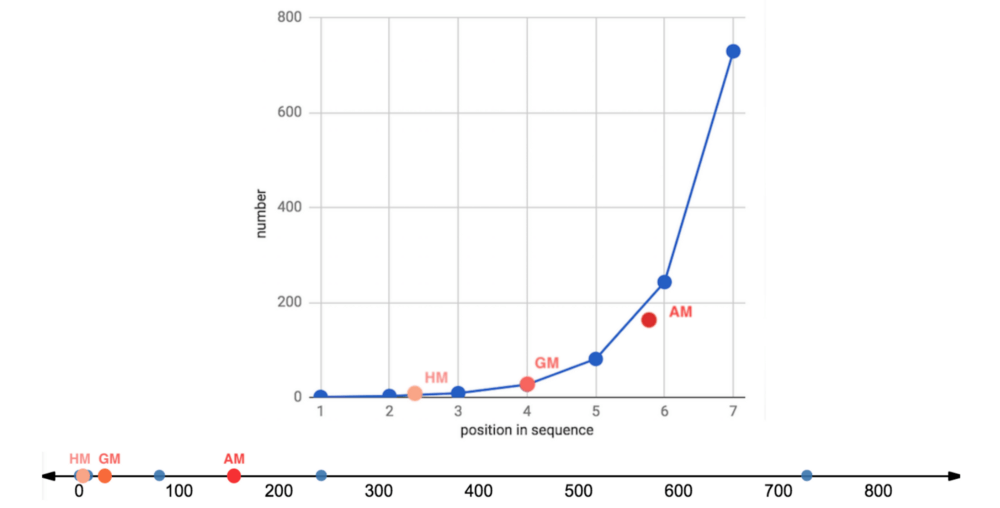
这篇则将在技术方面更深入一点,在一些概率分布合成的数据上探索这些平均数。接着我们将考察一些可供比较的“真实世界”大型数据集。这篇的使用的表述也会更简短,假定读者对高等数学和概率论有所了解。
毕达哥拉斯平均数温习
让我们复习一下,有3种毕达哥拉斯平均数,遵循如下不等关系:
调和平均数 ≤ 几何平均数 ≤ 算术平均数
仅当数据集中的所有数字都相等时,这3种平均数才相等。
算术平均数通过加法和除法得到。
几何平均数通过乘法和开方根得到。
调和平均数通过倒数、加法、除法得到。
它们的公式为:
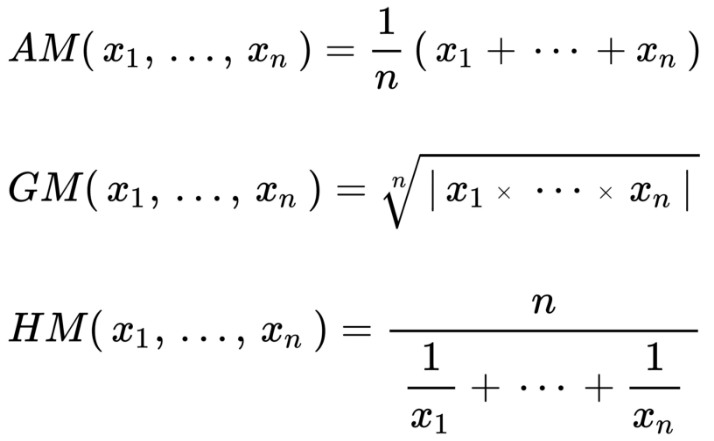
图片来源:维基百科
每种平均数可以表达为另一种平均数的再配置。例如:
几何平均数不过是数据集中的值的对数变换的算术平均数的反对数。有时它也能保留伸缩到同一分母后的算术平均数的次序。
调和平均数不过是数据集中的值的倒数的算术平均数的倒数。它也可以通过适当加权算术平均数重现。
经验规则:
算术平均数最适合相加的、线性的、对称的、正态/高斯数据集。
几何平均数最适合相乘的、几何的、指数的、对数正态分布的、扭曲的数据集,以及尺度不同的比率和复合增长的比率。
调和平均数是三种毕达哥拉斯平均数中最不常见的一种,但非常适合平均以分子为单位的比率,例如行程速度,一些财经指数,从物理到棒球的一些专门应用,还有评估机器学习模型。
限制:
由于相对不那么常用,几何平均数和调和平均数对一般受众而言可能难以理解甚至会误导他们。
几何平均数是无单位(unitless)的,尺度和可解释的单位在相乘操作中丢失了。
几何平均数和调和平均数无法处理包含0的数据集。
详细的讨论,请参阅上篇。下面我们将查看一些实际的例子。
合成数据集
上篇中,我们在一些微不足道的数据集(等差数列和等比数列)上观察了毕达哥拉斯平均数的效果。这里我们将查看一些更大的合成数据集(实数集上的多种概率分布)。
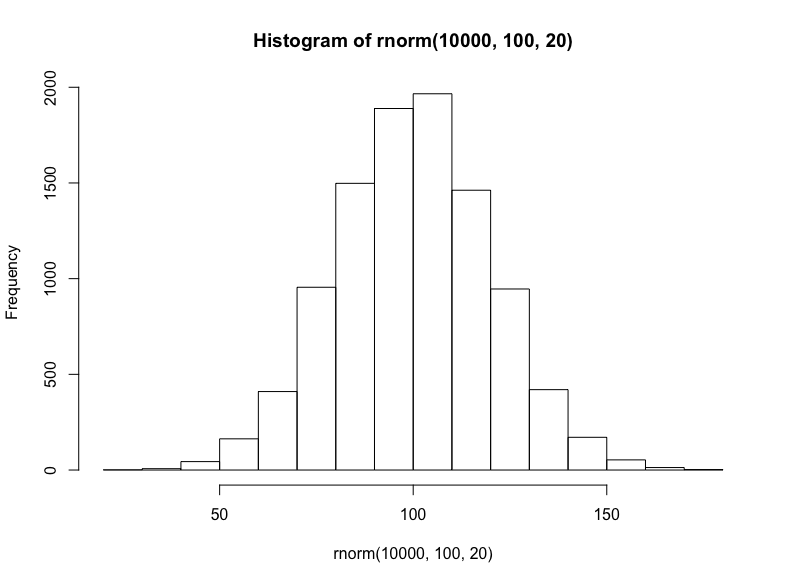
就相加或线性数据集而言,我们将从随机正态分布(均值100、标准差20)中抽取10000个样本:
hist(
rnorm( 10000, 100, 20 )
)
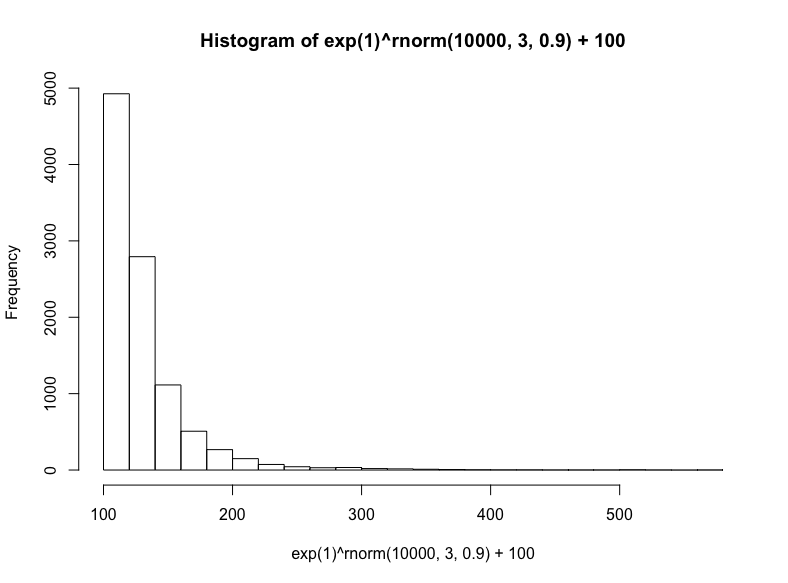
接着我们将模拟三种相乘数据集(尽管这些数据集具有有意义的差别,仍然常常难以区分):对数正态分布、指数分布、幂律分布。
有很多种生成对数正态分布的方法——基本上任何独立同分布的随机变量的乘法过程都将生成对数正态分布——这也正是它在真实世界中如此常见的原因,特别是在人类活动中。出于简单性和可解释性方面的考虑,我们将以欧拉数为底数,以从正态分布抽取的随机数为指数,然后加上100(使取值范围大致相当我们之前的正态分布):
hist(
exp(1)^rnorm(10000,3,.9) + 100,
breaks = 39
)
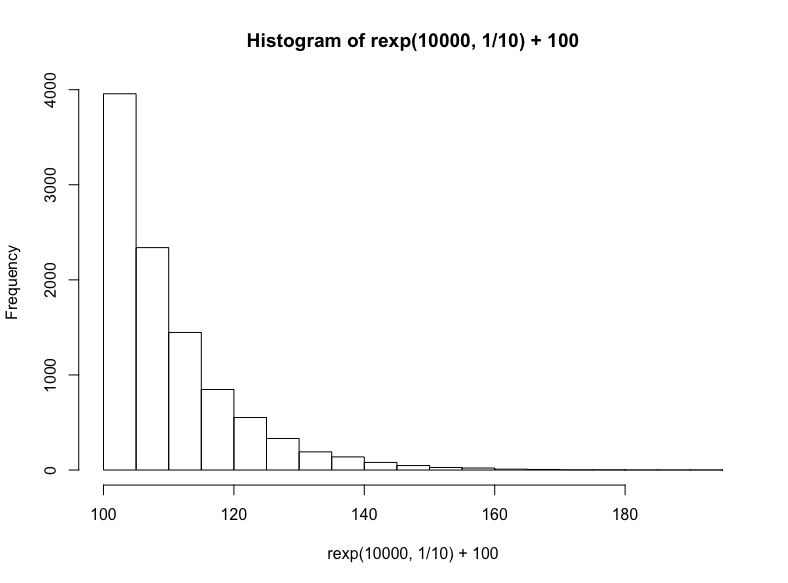
技术上说,这是指数分布的一个特例,但我们将通过R的rexp函数生成另一个指数分布,我们只需指定样本数以及衰减率(同样,我们在结果上加上100):
hist(
rexp(10000, 1/10) +100
)
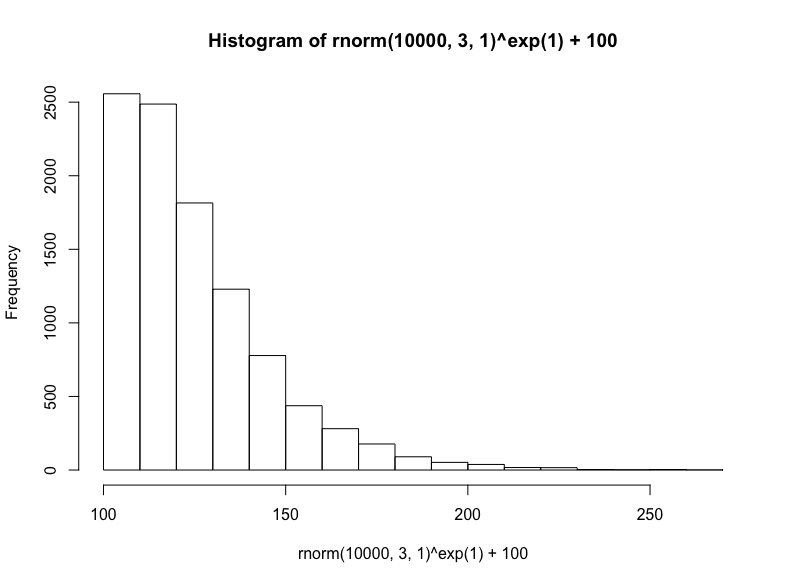
最后,我们将从正态分布取样底数,以欧拉数为指数,接着加上100,生成幂律分布:
(注意,这是对数正态方法的反向操作,在生成对数正态分布时,我们以欧拉数为底数,以正态分布取样为指数)
hist(
rnorm(10000, 3, 1)^exp(1) + 100
)
接着我们将使用ggridges包以更好地绘制分布,我们也将同时加载tidyverse包,任何有教养的R用户都这么干:
library(tidyverse)
library(ggridges)
dist1 <- rnorm(10000, 100, 20) %>%
tibble(x=., distribution = "normal")
dist2 <- ( exp(1)^rnorm(10000, 3, .9) + 100 ) %>%
tibble(x=., distribution = "lognormal")
dist3 <- ( rexp(10000, 1/10) + 100 ) %>%
tibble(x=., distribution = "exponential")
dist4 <- ( rnorm(10000,3,1)^exp(1) + 100 ) %>%
tibble(x=., distribution = "power law")
dists <- bind_rows(dist1, dist2, dist3, dist4)
dist_ord <- c("normal", "lognormal", "exponential", "power law")
dists <- dists %>%
mutate(distribution = fct_relevel(distribution, dist_ord))
ggplot(dists, aes(x = x, y = fct_rev(distribution), fill=..x..)) +
geom_density_ridges_gradient(quantiles = 2, scale=0.9,
color='white', show.legend = F) +
theme_minimal(base_size = 13, base_family = "sans") +
scale_y_discrete(expand = c(0.1, 0)) + xlim(0, 250) +
theme(panel.grid.major = element_line(colour = "white",
size = .3),
panel.grid.minor = element_blank(),
plot.background = element_rect(fill = "whitesmoke"),
axis.title = element_blank(), legend.position="none") +
ggtitle(label = "Distributions")
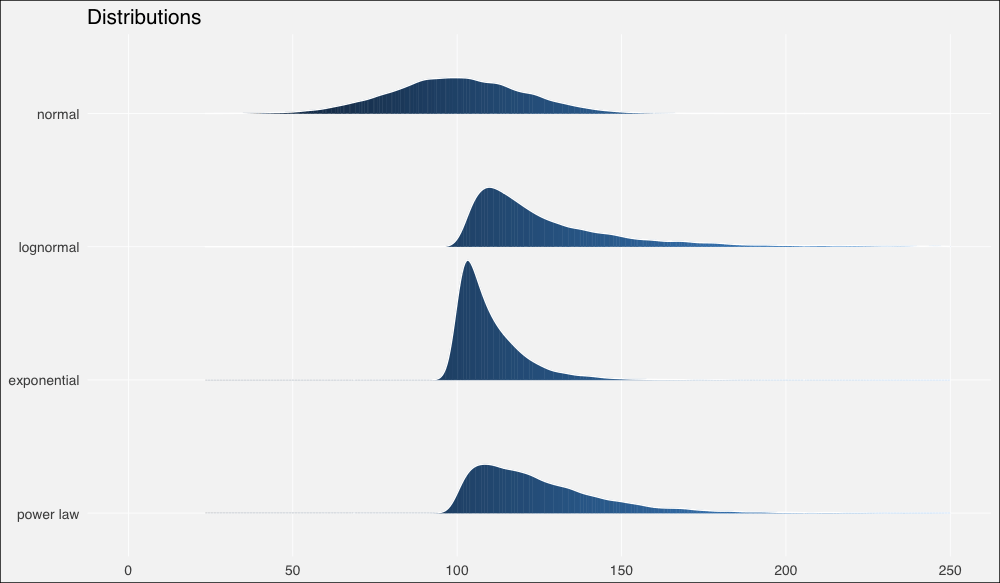
现在让我们计算一些概述统计量。
由于R没有内置几何平均数或调和平均数的函数,我们需要自行定义:
# 几何平均数函数
gm_mean = function(x, na.rm=TRUE){
exp(sum(log(x[x > 0]), na.rm=na.rm) / length(x))
}
dist_stats <- dists %>% group_by(distribution) %>%
summarise(median = median(x),
am = mean(x),
gm = gm_mean(x),
hm = 1/mean(1/x) # 调和平均数公式
)
输出:
# A tibble: 4 x 5
distribution median am gm hm
1 normal 99.699.997.795.4
2 lognormal 120 129 127 125
3 exponential 107 110 110 109
4 power law 120 125 124 122
……在绘制的图形上加上这些平均数:
ggplot(dists, aes(x = x, y = fct_rev(distribution), fill=..x..)) +
geom_density_ridges_gradient(quantiles = 2, scale=0.9,
color='white', show.legend = F) +
theme_minimal(base_size = 13, base_family = "sans") +
scale_y_discrete(expand = c(0.1, 0)) + xlim(0, 200) +
geom_point(data = dist_stats, aes(y=distribution, x=am),
colour="green3", shape=3, size=1, stroke =2,
alpha=.9, show.legend = F) +
geom_point(data = dist_stats, aes(y=distribution, x=gm),
colour="green3", fill="green3", shape=24, size=3,
alpha=.9, show.legend = F) +
geom_point(data = dist_stats, aes(y=distribution, x=hm),
colour="green3",fill= "green3", shape=25, size=3,
alpha=.9, show.legend = F) +
geom_segment(data = dist_stats, aes(x = median, xend = median,
y = c(4,3,2,1),
yend = c(4,3,2,1) + .3),
color = "salmon", show.legend = F) +
theme(panel.grid.major = element_line(colour = "white",
size = .3),
panel.grid.minor = element_blank(),
plot.background = element_rect(fill = "whitesmoke"),
axis.title = element_blank(), legend.position="none") +
ggtitle(label = "Distributions & summary statistics",
subtitle = "| : median : harmonic mean : geometric mean : arithmetic mean")
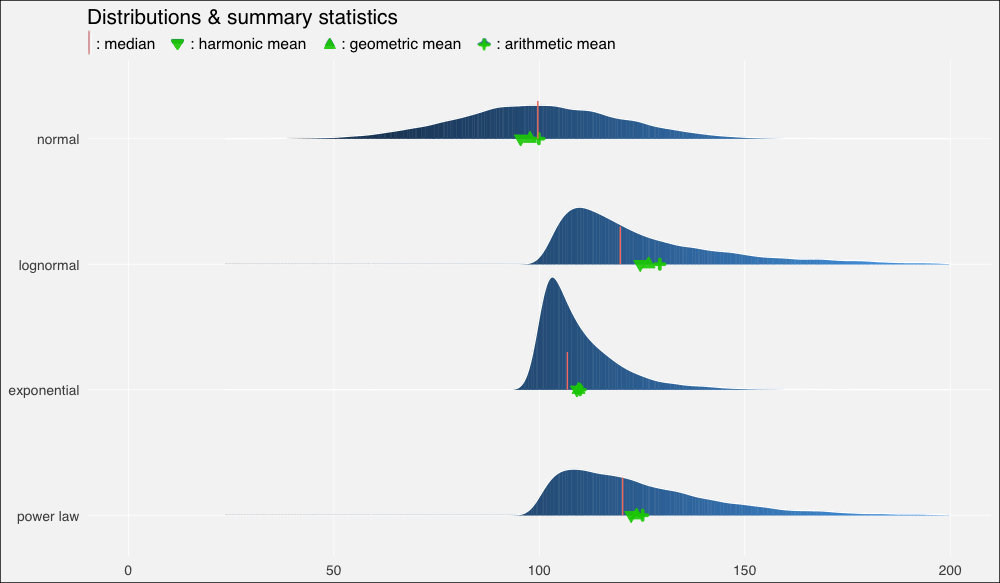
我们立刻看到了扭曲的密度的影响,以及平均数的重尾分布以及它们和中位数的关系:
在正态分布上,由于数据分布基本上是对称的,中位数和算术平均数几乎相等(分别是99.6和99.9)。
在向右扭曲的其他分布上,所有平均数均位于中位数右方,靠近数据集较密集的驼峰。
不过上图中的平均数有点拥挤,所以让我们放大一点来看(调整xlim()):
...xlim(90, 150)...
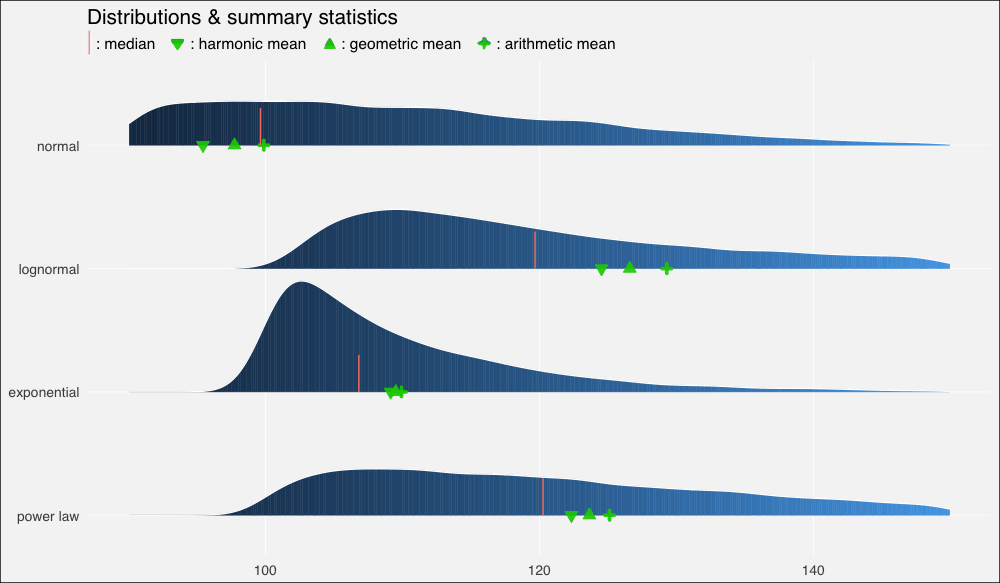
我们再一次看到,在正态、对称数据集上,几何平均数和调和平均数低估了数据的“中点”,不过三个平均数大致上间隔的空间是相等的。
在对数正态分布上,中等瘦削的长尾使平均数远离中位数,甚至也扭曲了平均数的分布,使算术平均数到几何平均数的距离比几何平均数到调和平均数的距离更远。
在指数分布上,数值高度密集,指数瘦削的短尾飞速衰减,使得平均数也挤作一团——尽管严重的扭曲仍然使它们偏离中位数。
幂律分布衰减较慢,也因此具有最肥的尾部。它的“主体”部分仍然是接近正态的,在不对称分布中的扭曲是最轻微的。平均数之间的距离大致相等,不过仍然远离中位数。
我之前提到过几何平均数和算术平均数之间的对数关系:
几何平均数不过是数据集中的值的对数变换的算术平均数的反对数。
为了验证这一点,让我们再看一看我们的概述统计量表格:
# A tibble: 4 x 5
distribution median am gm hm
1 normal 99.699.997.795.4
2 lognormal 120 129 127 125
3 exponential 107 110 110 109
4 power law 120 125 124 122
注意我们的对数正态分布的几何平均数是127.
现在我们计算对数变换值的算术平均数:
dist2$x %>% log() %>% mean()
输出:
4.84606
取反对数:
exp(1)^4.84606
输出:
127.2381
现在,为了把这一点讲透彻,让我们看看为什么会这样(以及对数正态是如何得名的):
减去我们原本加上的100,然后取对数:
(dist2$x — 100) %>% log() %>% hist()
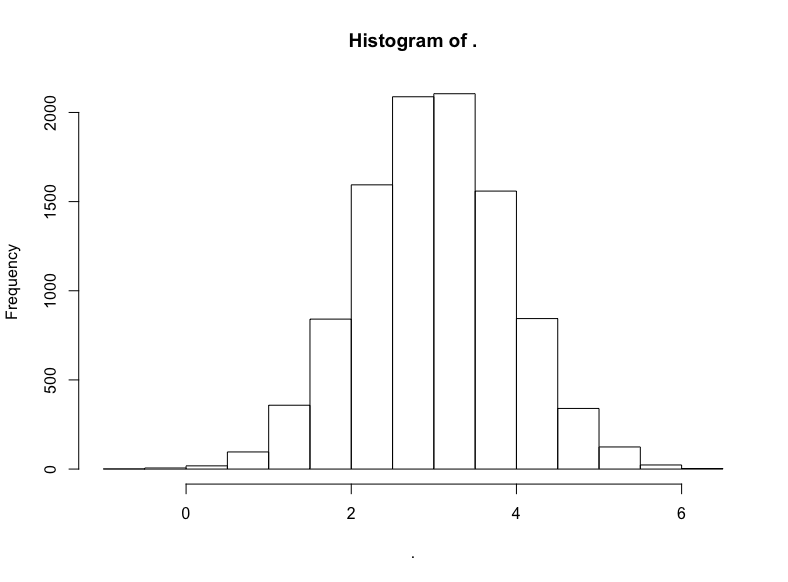
名副其实,对数正态分布的对数变换将产生正态分布。因此,正态分布中以相加为基础的算术平均数的结果与对数正态分布中以相乘为本质的几何平均数的结果是一致的。
我们不应该对对数正态分布的数据集经过对数变换得到的无可挑剔的正态分布过于印象深刻,毕竟我们在指定生成对数正态分布值的具体数据生成过程时就使用了正态分布,我们现在不过是反向操作以重现底层的正态分布而已。
现实世界中的事情很少如此整洁,现实世界的生成过程通常更复杂,是未知的,或者不可知的。因此如何建模和描述得自经验的数据集充满了困惑和争议。
让我们查看一些这样的数据集,了解下真实世界的烦恼。
真实世界数据
尽管通常不像模拟数据那样温顺,真实世界数据集通常至少再现了上述四种分布中的一种。
正态分布——喧闹的“钟形曲线”——最常出现在自然、生物场景中。身高和体重是经典的例子。因此,我们的第一直觉是看看可信赖的iris数据集。它确实满足要求,但样本数有点小(数据集中单种花卉的样本数为50)。我想要更大的数据集。
所以让我们加载bigrquery包:
library(bigrquery)
Google的BigQuery提供了众多真实数据的公开数据集,其中一些非常大,例如基因、专利、维基百科文章数据。
回到我们最初的目标,natality(译者注:natality意为出生率)看起来就很生物:
project <- “YOUR-PROJECT-ID”
sql <- “SELECT COUNT(*)
FROM `bigquery-public-data.samples.natality`”
query_exec(sql, project = project, use_legacy_sql = F)
(提示:由于有海量数据,因此你可能需要为访问数据付费,不过每个月前1TB的数据访问是免费的。另外,尽管出于明显的原因,强烈不推荐使用SELECT *,SELECT COUNT(*)却是一项免费的操作,使用它确定范围是个好主意。)
输出:
137826763
一亿三千七百万婴儿数据!我们用不了这么多,所以让我们随机取样1%婴儿的体重,获取前一百万结果:
sql <- “SELECT weight_pounds
FROM `bigquery-public-data.samples.natality`
WHERE RAND() < .01”
natal <- query_exec(sql, project = project, use_legacy_sql = F,
max_pages = 100)
hist(natal$weight_pounds)生成:
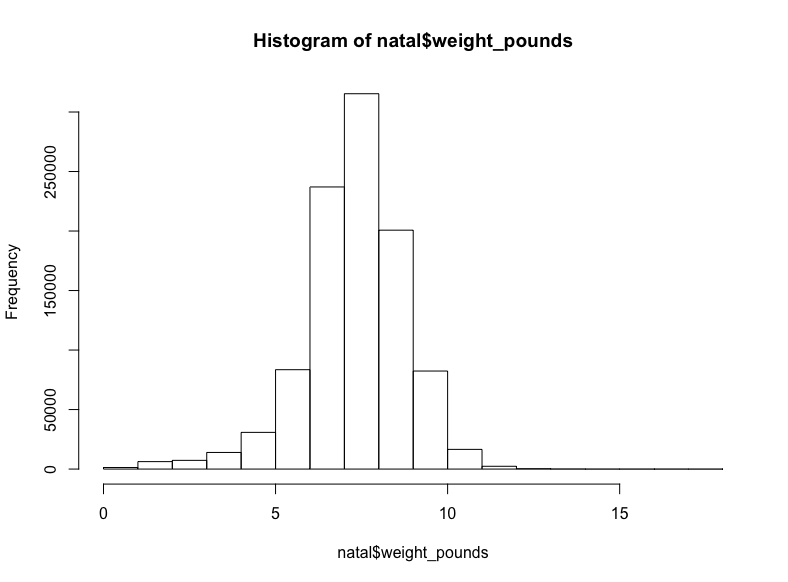
至少在我看来这是正态分布。
现在让我们找找有些扭曲的相乘数据,让我们从生物学转向社会学。
我们将查看New York(纽约)数据集,其中包含各种城市信息,包括黄色出租车和绿色出租车的行程信息(译者注:纽约的出租车分为黄、绿两种,两者允许接客区域不同)。
sql <- “SELECT COUNT(*)
FROM `nyc-tlc.green.trips_2015`”
query_exec(sql, project = project, use_legacy_sql = F)
输出:
9896012
不到一千万条记录,所以让我们抓取所有的行程距离:
(这可能需要花费一点时间)
sql <- "SELECT trip_distance FROM `nyc-tlc.green.trips_2015`"
trips <- query_exec(sql, project = project, use_legacy_sql = F)
hist(trips$trips_distance)
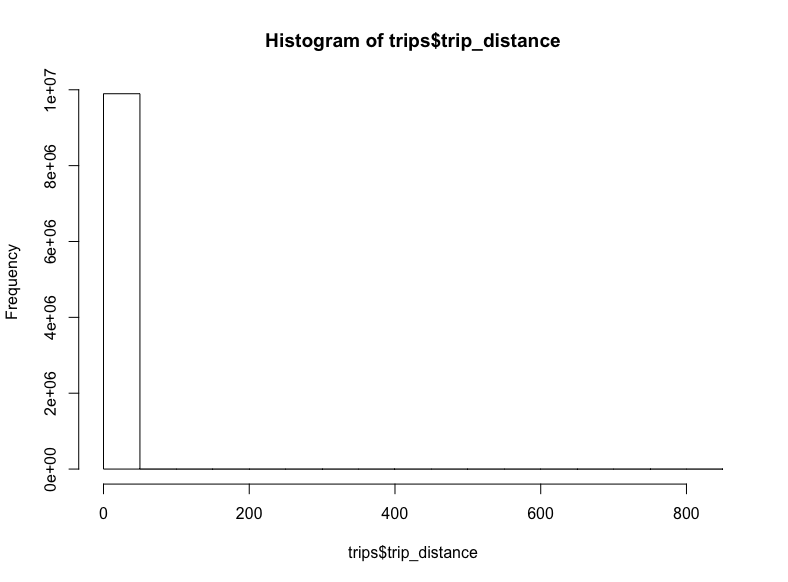
-_-
看起来一些极端的离散值将我们的x轴拉到了八百英里开外。对出租车而言,这也太远了。让我们移除这些离散值,将距离限定至20英里:
trips$trip_distance %>% subset(. <= 20) %>% hist()
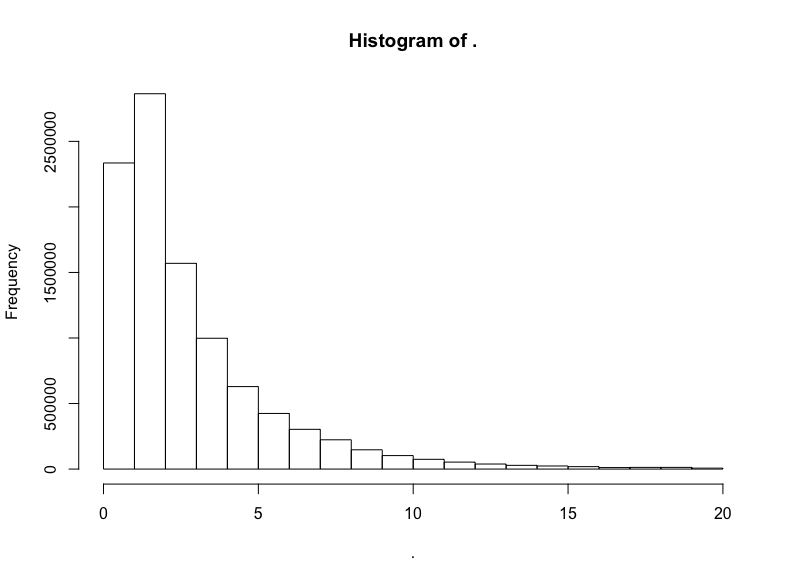
我们做到了,得到了对数正态分布标志性的长尾。让我们验证一下分布的对数正态性,绘制对数的直方图:
trips$trip_distance %>% subset(. <= 20) %>% log() %>% hist()
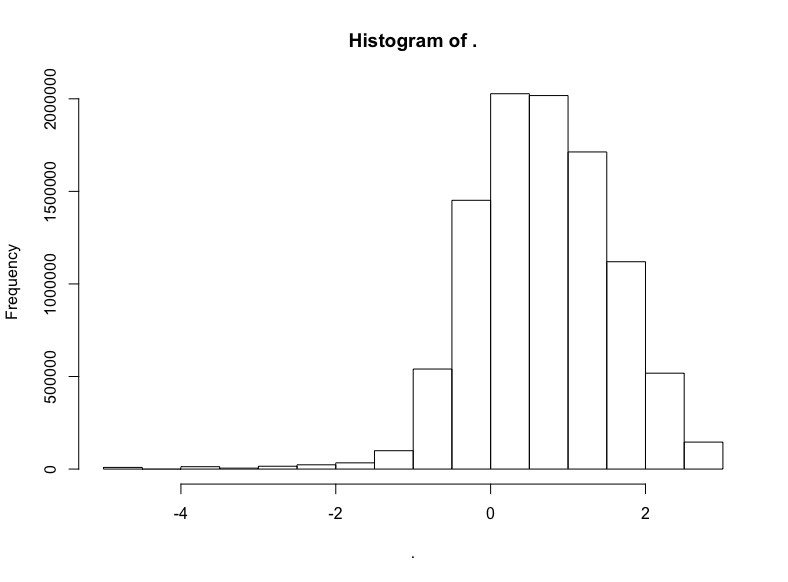
明显有正态分布的样子,不过偏离了一点靶心,有一点向左扭曲。哎呀,真实世界就是这样的。不过我们有把握说,应用对数正态分布至少不算荒谬。
让我们继续前行。寻找更重尾分布的数据。这次我们将使用Github数据集:
sql <- "SELECT COUNT(*)
FROM `bigquery-public-data.samples.github_nested`"
query_exec(sql, project = project, use_legacy_sql = F)
输出:
2541639
二百五十万项记录。我开始为本地机器的内存担心了,所以我将通过随机取样去掉一半数据,然后查看剩余代码仓库的关注数(watchers):
sql <- “SELECT repository.watchers
FROM `bigquery-public-data.samples.github_nested`
WHERE RAND() < .5”
github <- query_exec(sql, project = project, use_legacy_sql = F,
max_pages = 100)
github$watchers %>% hist()
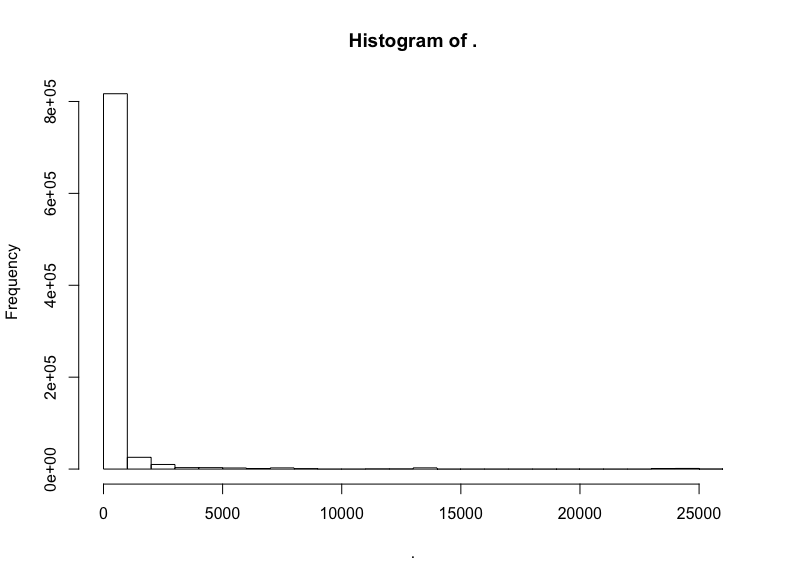
极端的长尾,所以让我们移除过低和过高的关注数:
github$watchers %>% subset(5 < . & . < 3000) %>% hist()
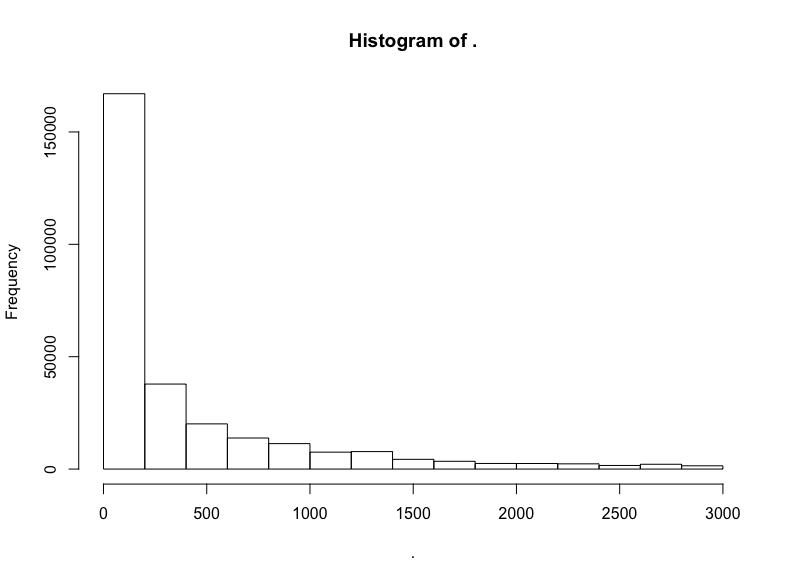
这是指数分布。
但是它是不是同时也是对数正态分布?
github$watchers %>% subset(5 < . & . < 3000) %>% log() %>% hist()
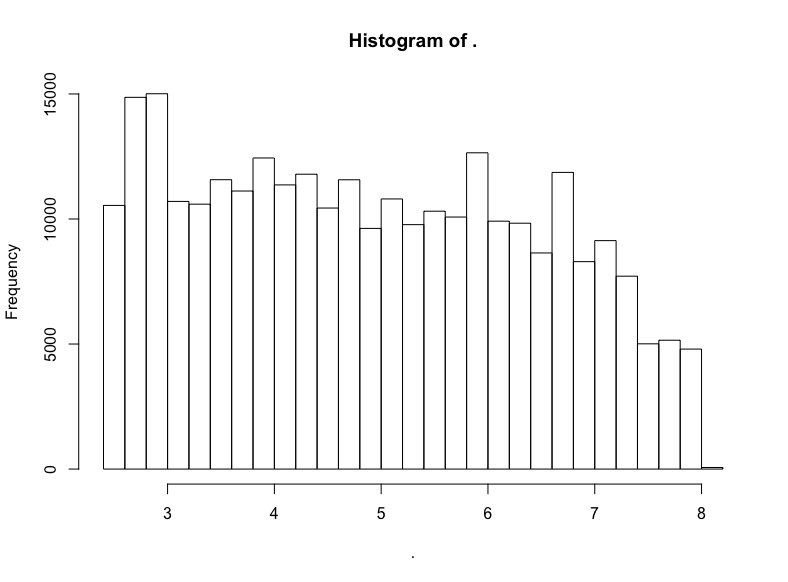
否。
不过我们看到了一头珍稀的野兽:(逼近)LogUniform分布!
让我们再从大数据中抽取一次,这次我们将查看Hacker News帖子的评分:
sql <- “SELECT COUNT(*)
FROM `bigquery-public-data.hacker_news.full`”
query_exec(sql, project = project, use_legacy_sql = F)
输出:
16489224
我们抽取前10%的样本:
sql <- “SELECT score
FROM `bigquery-public-data.hacker_news.full`
WHERE RAND() < .1”
hn <- query_exec(sql, project = project, use_legacy_sql = F,
max_pages = 100)
hn$score %>% hist()
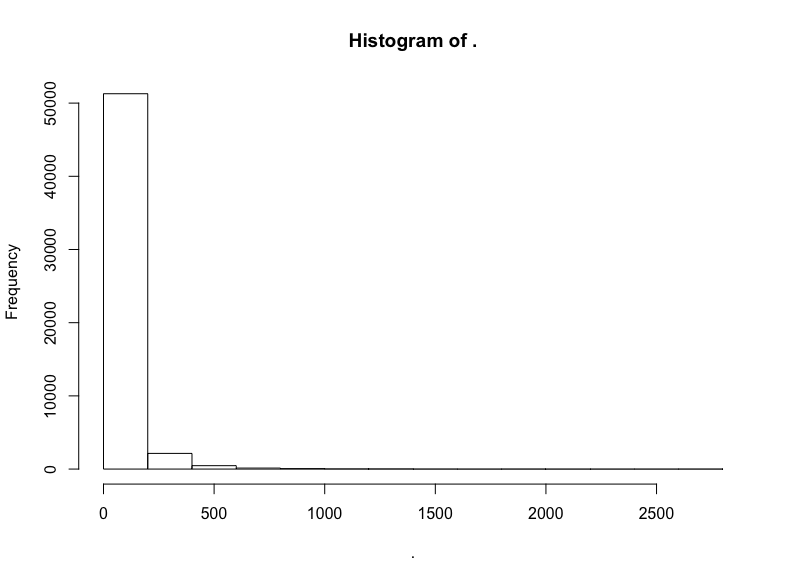
同样,我们截取中间部分的评分:
hn$score %>% subset(10 < . & . <= 300) %>% hist()
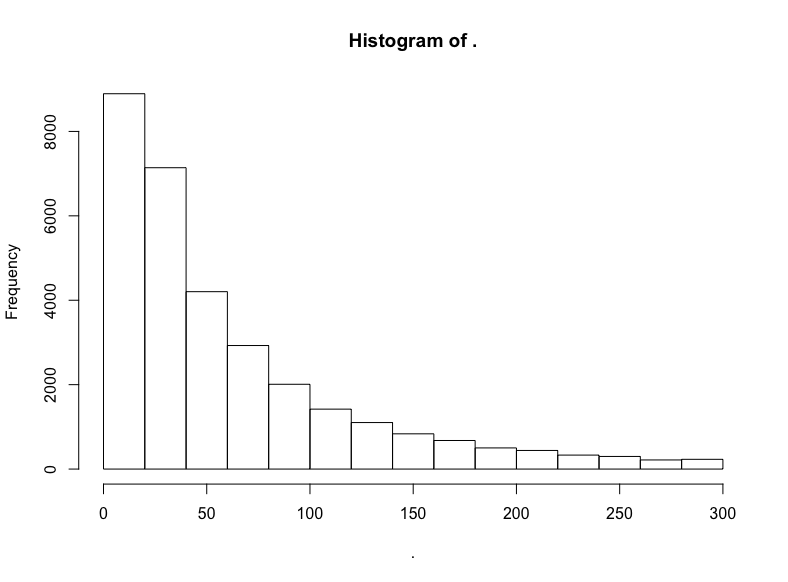
截取中间部分后,衰减得慢了。看看对数变换的结果?
hn$score %>% subset(10 < . & . <= 300) %>% log() %>% hist()
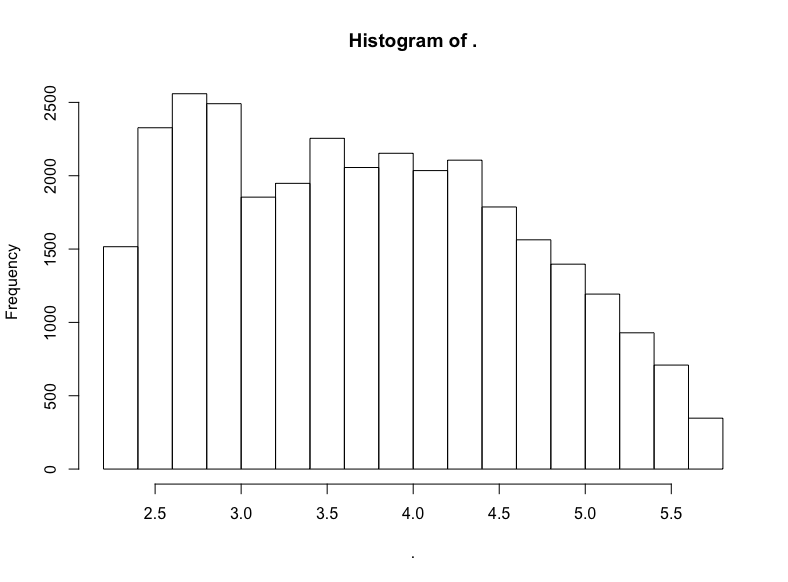
同样大致是右向衰减的LogUniform分布。
我对幂律分布的搜寻没有得到结果,这也许并不值得惊讶,毕竟幂律分布最常出现在网络科学中(甚至,即使在网络科学中,幂律分布看起来也比最初宣称的要罕见)。
不管怎么说,让我们也像模拟分布那样绘制真实数据集的分布图,并加以对比。同样,我们将对其加以标准化,使其位于100左右。
# 定制标准化函数
normalize = function(x, na.rm = T){
(x-min(x[!is.na(x)]))/(max(x[!is.na(x)])-min(x[!is.na(x)]))
}
rndist1 <- (normalize(natality$weight_pounds) + 100) %>%
tibble(x=., distribution = "natal weights")
trip_trim <- trips$trip_distance %>% subset(. <= 20)
rndist2 <- (normalize(trip_trim) + 100) %>%
tibble(x=., distribution = "nyc green cab trips")
git_trim <- github$watchers %>% subset(5 < . & . < 3000)
rndist3 <- (normalize(git_trim) + 100) %>%
tibble(x=., distribution = "github watchers")
hn_trim <- hn$score %>% subset(10 < . & . <= 300)
rndist4 <- (normalize(hn_trim) + 100) %>%
tibble(x=., distribution = "hacker news scores")
rndists <- bind_rows(rndist1, rndist2, rndist3, rndist4)
rndist_ord <- c("natal weights", "nyc green cab trips",
"github watchers", "hacker news scores")
rndists <- rndists %>%
mutate(distribution = fct_relevel(distribution, rndist_ord))
ggplot(rndists, aes(x = x, y = fct_rev(distribution), fill=..x..)) +
geom_density_ridges_gradient(quantiles = 2, scale=0.9,
color='white', show.legend = F) +
theme_minimal(base_size = 13, base_family = "sans") +
scale_y_discrete(expand = c(0.1, 0)) + xlim(99.5, 101) +
theme(panel.grid.major = element_line(colour = "white", size = .3),
panel.grid.minor = element_blank(),
plot.background = element_rect(fill = "whitesmoke"),
axis.title = element_blank(), legend.position="none") +
ggtitle(label = "Distributions")
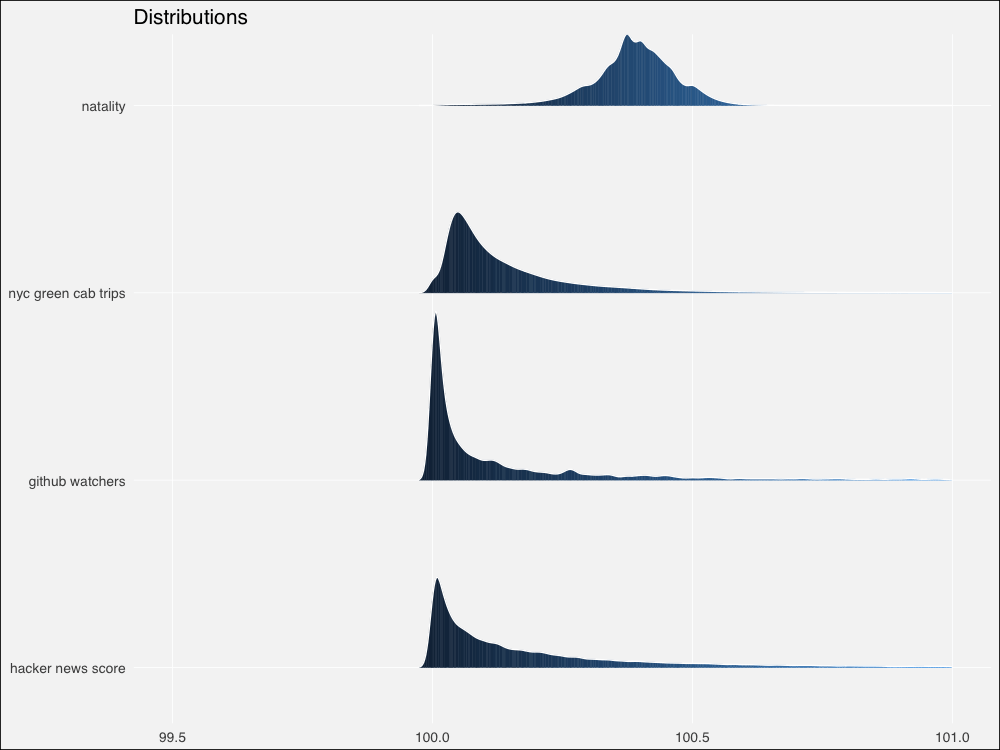
由于来自真实世界,和模拟分布相比,边缘更加粗糙不平。不过仍然看起来相似。让我们用Thomas Lin Pedersen的patchwork包绘制模拟分布和真实分布的对比图:
# 将图形分配给对象
p1 <-ggplot(dists, aes(x = x, y = fct_rev(distribution), fill=..x..)) +
geom_density_ridges_gradient(quantiles = 2, scale=0.9,
color='white', show.legend = F) +
theme_minimal(base_size = 13, base_family = "sans") +
scale_y_discrete(expand = c(0.1, 0)) + xlim(0, 250) +
theme(panel.grid.major = element_line(colour = "white", size = .3),
panel.grid.minor = element_blank(),
plot.background = element_rect(fill = "whitesmoke"),
axis.title = element_blank(), legend.position="none") +
ggtitle(label = "Distributions")
p2 <- ggplot(rndists, aes(x = x, y = fct_rev(distribution), fill=..x..)) +
geom_density_ridges_gradient(quantiles = 2, scale=0.9,
color='white', show.legend = F) +
theme_minimal(base_size = 13, base_family = "sans") +
scale_y_discrete(expand = c(0.1, 0)) + xlim(99.5, 101) +
theme(panel.grid.major = element_line(colour = "white", size = .3),
panel.grid.minor = element_blank(),
plot.background = element_rect(fill = "whitesmoke"),
axis.title = element_blank(), legend.position="none") +
ggtitle(label = "Distributions")
# 直接将图形对象相加
p1 + p2
总体来看,模拟分布将为相邻的真实数据集提供合理的模型,除了幂律 -> HackerNews评分这一对例外。
当然有很多模型拟合方面的严谨测试,不过让我们直接在分布上绘制概述统计量,就像我们之前做的那样。
不幸的是,标准化的真实世界数据扭曲了概述统计量计算,使得结果多多少少难以区分。我怀疑这可能是因为计算机的浮点计算精度问题(不过也可能只是因为我自己在数值计算上犯了错误)。我不得不使用未标准化的真实世界数据单独绘制,然后尝试手工对齐。
让我们组合未标准化的分布,然后计算概述性数据:
# 未标准化
rdist1 <- natality$weight_pounds %>%
tibble(x=., distribution = "natal weights")
rdist2 <- trips$trip_distance %>% subset(. <= 20) %>%
tibble(x=., distribution = "nyc green cab trips")
rdist3 <- github$watchers %>% subset(5 < . & . < 3000) %>%
tibble(x=., distribution = "github watchers")
rdist4 <- hn$score %>% subset(10 < . & . <= 300) %>%
tibble(x=., distribution = "hacker news scores")
rdists <- bind_rows(rdist1, rdist2, rdist3, rdist4)
rdist_ord <- c("natal weights", "nyc green cab trips",
"github watchers", "hacker news scores")
rdists <- rdists %>%
mutate(distribution = fct_relevel(distribution, rdist_ord))
rdist_stats <- rdists %>% group_by(distribution) %>%
summarise(median = median(x, na, na.rm = T),
am = mean(x, na.rm = T),
gm = gm_mean2(x[x>0]),
hm = 1/mean(1/x[x>0], na.rm = T))
现在让我们绘图(这很丑陋因为我们需要为每个真实分布创建单独的图形,好在patchwork让我们可以优雅地定义布局):
# 模拟分布的绘图和之前一样
pm1 <- ggplot(dists, aes(x = x, y = fct_rev(distribution), fill=..x..)) +
geom_density_ridges_gradient(quantiles = 2, scale=0.9,
color='white', show.legend = F) +
theme_minimal(base_size = 13, base_family = "sans") +
scale_y_discrete(expand = c(0.1, 0)) + xlim(0, 250) +
geom_point(data = dist_stats, aes(y=distribution, x=am),
colour="green3", shape=3, size=1, stroke =2,
alpha=.9, show.legend = F) +
geom_point(data = dist_stats, aes(y=distribution, x=gm),
colour="green3", fill="green3", shape=24, size=3,# stroke = 1,
alpha=.9, show.legend = F) +
geom_point(data = dist_stats, aes(y=distribution, x=hm),
colour="green3",fill= "green3", shape=25, size=3,# stroke = 1,
alpha=.9, show.legend = F) +
geom_segment(data = dist_stats, aes(x = median, xend = median,
y = c(4,3,2,1),
yend = c(4,3,2,1) + .3),
color = "salmon", show.legend = F)+
theme(panel.grid.major = element_line(colour = "white", size = .3),
panel.grid.minor = element_blank(),
plot.background = element_rect(fill = "whitesmoke"),
axis.title = element_blank(), legend.position="none") +
ggtitle(label = "Distributions & summary statistics",
subtitle = "| : median : harmonic mean : geometric mean : arithmetic mean")
# 真实数据
p3 <- ggplot(rdist1, aes(x = x, y = distribution, fill = ..x..)) +
geom_density_ridges_gradient(quantiles = 2, scale=0.9,
color='white', show.legend = F) +
theme_minimal(base_size = 13, base_family = "sans") +
scale_y_discrete(expand = c(0.1, 0)) + #xlim(3, 11) +
geom_point(data = rdist_stats[1,], aes(y=distribution, x=am),
colour="green3", shape=3, size=1, stroke =2,
alpha=.9, show.legend = F) +
geom_point(data = rdist_stats[1,], aes(y=distribution, x=gm),
colour="green3", fill="green3", shape=24, size=3,# stroke = 1,
alpha=.9, show.legend = F) +
geom_point(data = rdist_stats[1,], aes(y=distribution, x=hm),
colour="green3",fill= "green3", shape=25, size=3,# stroke = 1,
alpha=.9, show.legend = F) +
geom_segment(data = rdist_stats[1,], aes(x = median, xend = median,
y = 1,
yend = 1 + .3),
color = "salmon", show.legend = F)+
theme(panel.grid.major = element_line(colour = "white", size = .3),
panel.grid.minor = element_blank(),
plot.background = element_rect(fill = "whitesmoke"),
axis.title = element_blank(), legend.position="none",
axis.text.x=element_blank())
p4 <- ggplot(rdist2, aes(x = x, y = distribution, fill = ..x..)) +
geom_density_ridges_gradient(quantiles = 2, scale=0.9,
color='white', show.legend = F) +
theme_minimal(base_size = 13, base_family = "sans") +
scale_y_discrete(expand = c(0.1, 0)) + #xlim(-1, 4) +
geom_point(data = rdist_stats[2,], aes(y=distribution, x=am),
colour="green3", shape=3, size=1, stroke =2,
alpha=.9, show.legend = F) +
geom_point(data = rdist_stats[2,], aes(y=distribution, x=gm),
colour="green3", fill="green3", shape=24, size=3,# stroke = 1,
alpha=.9, show.legend = F) +
geom_point(data = rdist_stats[2,], aes(y=distribution, x=hm),
colour="green3",fill= "green3", shape=25, size=3,# stroke = 1,
alpha=.9, show.legend = F) +
geom_segment(data = rdist_stats[2,], aes(x = median, xend = median,
y = 1,
yend = 1 + .3),
color = "salmon", show.legend = F)+
theme(panel.grid.major = element_line(colour = "white", size = .3),
panel.grid.minor = element_blank(),
plot.background = element_rect(fill = "whitesmoke"),
axis.title = element_blank(), legend.position="none",
axis.text.x=element_blank())
p5 <- ggplot(rdist3, aes(x = x, y = distribution, fill = ..x..)) +
geom_density_ridges_gradient(quantiles = 2, scale=200,
color='white', show.legend = F) +
theme_minimal(base_size = 13, base_family = "sans") +
scale_y_discrete(expand = c(0.1, 0)) + #xlim(20, 450) +
geom_point(data = rdist_stats[3,], aes(y=distribution, x=am),
colour="green3", shape=3, size=1, stroke =2,
alpha=.9, show.legend = F) +
geom_point(data = rdist_stats[3,], aes(y=distribution, x=gm),
colour="green3", fill="green3", shape=24, size=3,# stroke = 1,
alpha=.9, show.legend = F) +
geom_point(data = rdist_stats[3,], aes(y=distribution, x=hm),
colour="green3",fill= "green3", shape=25, size=3,# stroke = 1,
alpha=.9, show.legend = F) +
geom_segment(data = rdist_stats[3,], aes(x = median, xend = median,
y = 1,
yend = 1 + .3),
color = "salmon", show.legend = F)+
theme(panel.grid.major = element_line(colour = "white", size = .3),
panel.grid.minor = element_blank(),
plot.background = element_rect(fill = "whitesmoke"),
axis.title = element_blank(), legend.position="none",
axis.text.x=element_blank())
p6 <- ggplot(rdist4, aes(x = x, y = distribution, fill = ..x..)) +
geom_density_ridges_gradient(quantiles = 2, scale=40,
color='white', show.legend = F) +
theme_minimal(base_size = 13, base_family = "sans") +
scale_y_discrete(expand = c(.01, 0)) + #xlim(0, 100) +
geom_point(data = rdist_stats[4,], aes(y=distribution, x=am),
colour="green3", shape=3, size=1, stroke =2,
alpha=.9, show.legend = F) +
geom_point(data = rdist_stats[4,], aes(y=distribution, x=gm),
colour="green3", fill="green3", shape=24, size=3,# stroke = 1,
alpha=.9, show.legend = F) +
geom_point(data = rdist_stats[4,], aes(y=distribution, x=hm),
colour="green3",fill= "green3", shape=25, size=3,# stroke = 1,
alpha=.9, show.legend = F) +
geom_segment(data = rdist_stats[4,], aes(x = median, xend = median,
y = 1,
yend = 1 + .3),
color = "salmon", show.legend = F)+
theme(panel.grid.major = element_line(colour = "white", size = .3),
panel.grid.minor = element_blank(),
plot.background = element_rect(fill = "whitesmoke"),
axis.title = element_blank(), legend.position="none")
# 魔法般的patchwork布局语法
pm1 | (p3 / p4 / p5 / p6)
有趣的是,我们的真实世界数据集的概述统计量看起来明显比模拟分布上更为分散。让我们放大一点看看。
为了节省篇幅,我这里不会重复粘贴代码,基本上我不过是修改了pm1的xlim(90, 150),并且去掉了上述代码中的xlim()行的注释:
放大后对比更鲜明了。
我们对模拟和真实世界分布上的毕达哥拉斯平均数的探索到此为止。
如果你还没有看过上篇,可以看一下,上篇给出了一个更明确、更直观的介绍。同时,别忘了参考后面给出的链接和进一步阅读。
另外,如果你想读到更多这样的文章,可以在Twitter、LinkedIn、Github上关注我(我在上面的用户名都是dnlmc)。
-
新手求助:请问怎么将数组中每100个数,求平均数2015-03-06 12698
-
新手求助:请问怎么将数组中每隔n个数平均数2015-03-07 6844
-
怎么查询平均数2018-11-13 1128
-
如何实现c++自动输出平均数?2019-10-10 1740
-
求无符号数的平均数2021-12-01 1042
-
如何操作多台过采样数据转换器的详细资料概述2018-05-29 1094
-
LabVIEW在信捷PLC通讯上的应用详细资料概述2018-06-07 3458
-
如何在开发板上实现交通灯模拟的详细资料概述2018-06-19 1060
-
几何平均数和调和平均数是什么?有什么作用?详细资料讨论2018-07-19 55783
-
SV601187的详细资料合集包括了电路图,原理图和介绍等详细资料概述2018-07-30 1716
-
组态王与数据库连接的实现方法详细资料概述2018-10-31 3967
-
python的内置函数详细资料概述2019-11-18 1185
-
CAN总线基础的详细资料概述2019-11-29 2174
-
EMC HF垫圈的详细资料概述2020-09-07 1310
-
ADRF5144: 10 W平均数,硅SPDT,反射开关,1千赫至20千兆赫数据表 ADI2023-10-11 56
全部0条评论

快来发表一下你的评论吧 !

