
资料下载

工程微生物相互作用的工具包
描述
项目概况
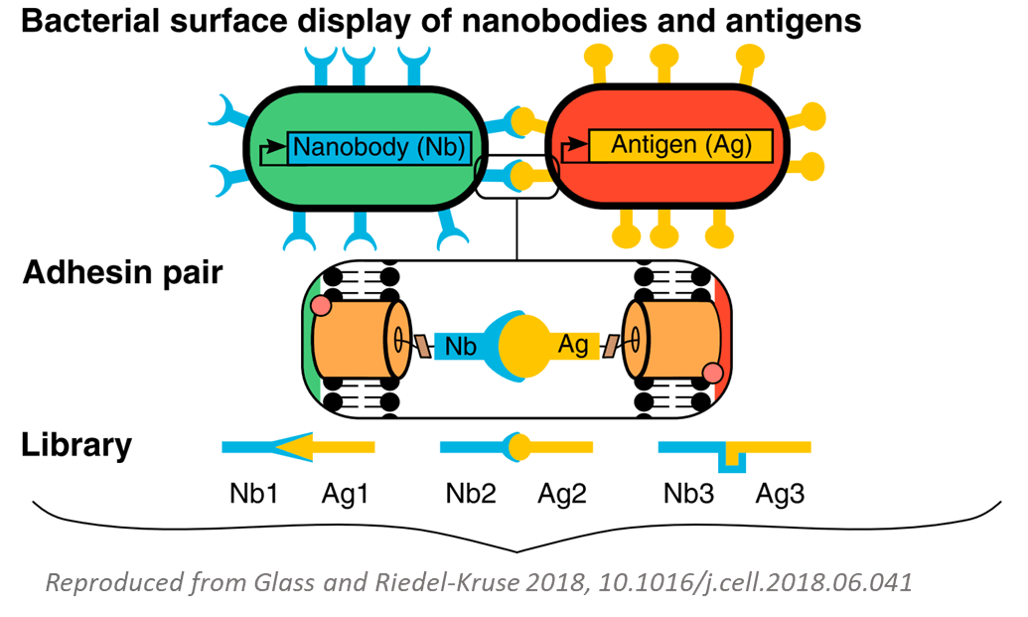
复杂群落中细胞之间的相互作用会导致出现超出简单细胞单一培养所能达到的意想不到的功能行为。在利用细胞群落推进一系列合成生物学应用(从生物材料到更高效的细胞工厂)方面存在巨大的未开发潜力。我们正在构建一个工具包,用于系统地设计两种细胞类型的混合种群之间可能发生的结构和生态相互作用。我们的工具包将提供两种人工表面受体,用于构建大肠杆菌之间粘附相互作用的组合以及用于在细胞之间产生协同和对抗生态关系的模块化电路。借助这组相互兼容且具有功能特征的部件,我们将为科学家、教育工作者和基于社区的实验室提供开放且可访问的资源,以开始对多细胞细菌群落进行编程。
生物积木
为了设计具有明确相互作用的细菌系统,我们利用了 3 个主要的生物“构建块”。这些是 1) 编码蛋白质的合成基因,这些蛋白质允许细胞选择性地识别和结合其他细胞(称为“纳米抗体”),2)不能自行产生某些必需营养素的基因突变细菌菌株(称为“营养缺陷型”), 3) 称为质粒的便携式 DNA 元件,可产生抗菌毒素并对这些毒素产生选择性免疫。
纳米抗体粘附受体
我们有几种用质粒转化的细菌菌株,这些质粒编码纳米抗体-抗原对。这些纳米抗体蛋白在已用适当质粒成功转化的细菌细胞表面表达。其他细菌细胞如果用合适的质粒转化,它们的细胞表面会显示抗原。
纳米抗体将以高特异性和亲和力结合特定的表面抗原,因此显示互补纳米抗体-抗原对的不同细胞将相互粘附并形成团块。这些细胞还标记有不同的荧光蛋白,因此可以在显微镜下检测分析新兴结构。
营养缺陷型突变体
我们有 8个大肠杆菌突变体,它们对 4 种不同的氨基酸(色氨酸、甲硫氨酸、脯氨酸和异亮氨酸)呈营养缺陷型,并带有 eGFP 或 mCherry 标记。
每个营养缺陷型将无法生长,除非它所在的培养基补充了相关的氨基酸,并且这些营养缺陷型的生长可以通过它们的荧光(在显微镜下的读板器中)或通过观察浊度来检测(在读板器或分光光度计中)。
氨基酸会扩散出大肠杆菌细胞,因此同一环境中的不同营养缺陷型可能能够彼此共享资源,从而在没有补充氨基酸的培养基中维持生长。这可用于生成互惠交互对。
毒素-抗毒素系统
细菌已经进化出可以分泌毒素的自然系统,毒素是用来杀死其他细胞的大分子。可以用编码产生毒素-抗毒素对的质粒转化细菌,从而赋予产生毒素的细菌对毒素的抗性。这些释放到周围介质中的毒素会伤害没有抗毒素的其他细菌,从而在细胞之间产生对抗性的社会相互作用。
结果(截至2019.10.30)
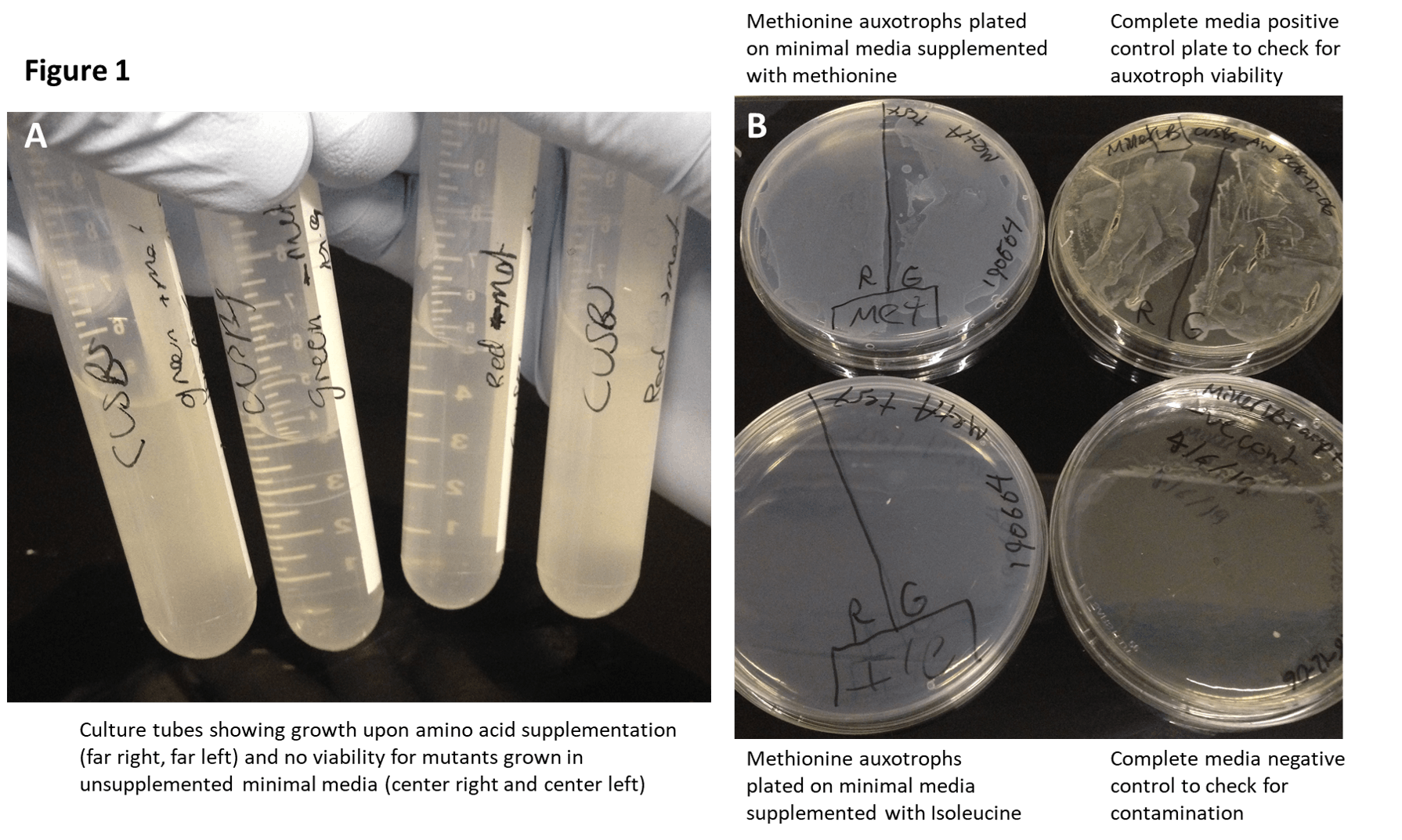
设置突变菌株的生长条件
我们首先必须调整生长条件,以便营养缺陷型突变体大肠杆菌在我们的工具包中将无法自行增长,除非媒体得到补充。在碳源有限的严格培养基配方上未能看到任何生长后,我们在添加 0.4% 葡萄糖的 M9 基本培养基中看到了合理的生长。我们可以通过补充过量(即 100 ug/mL)各自的氨基酸来挽救蛋氨酸、异亮氨酸、脯氨酸和色氨酸营养缺陷型的生长(图 1A)。在补充和未补充的平板上,我们证实其他氨基酸不能替代我们的任何营养缺陷型,并且观察到的生长不仅仅是来自污染(图 1B)。为了观察这些影响,我们必须接种相对少量的细菌 (<3 uL)。更高的接种量导致突变体在所有培养基条件下的非特异性生长,
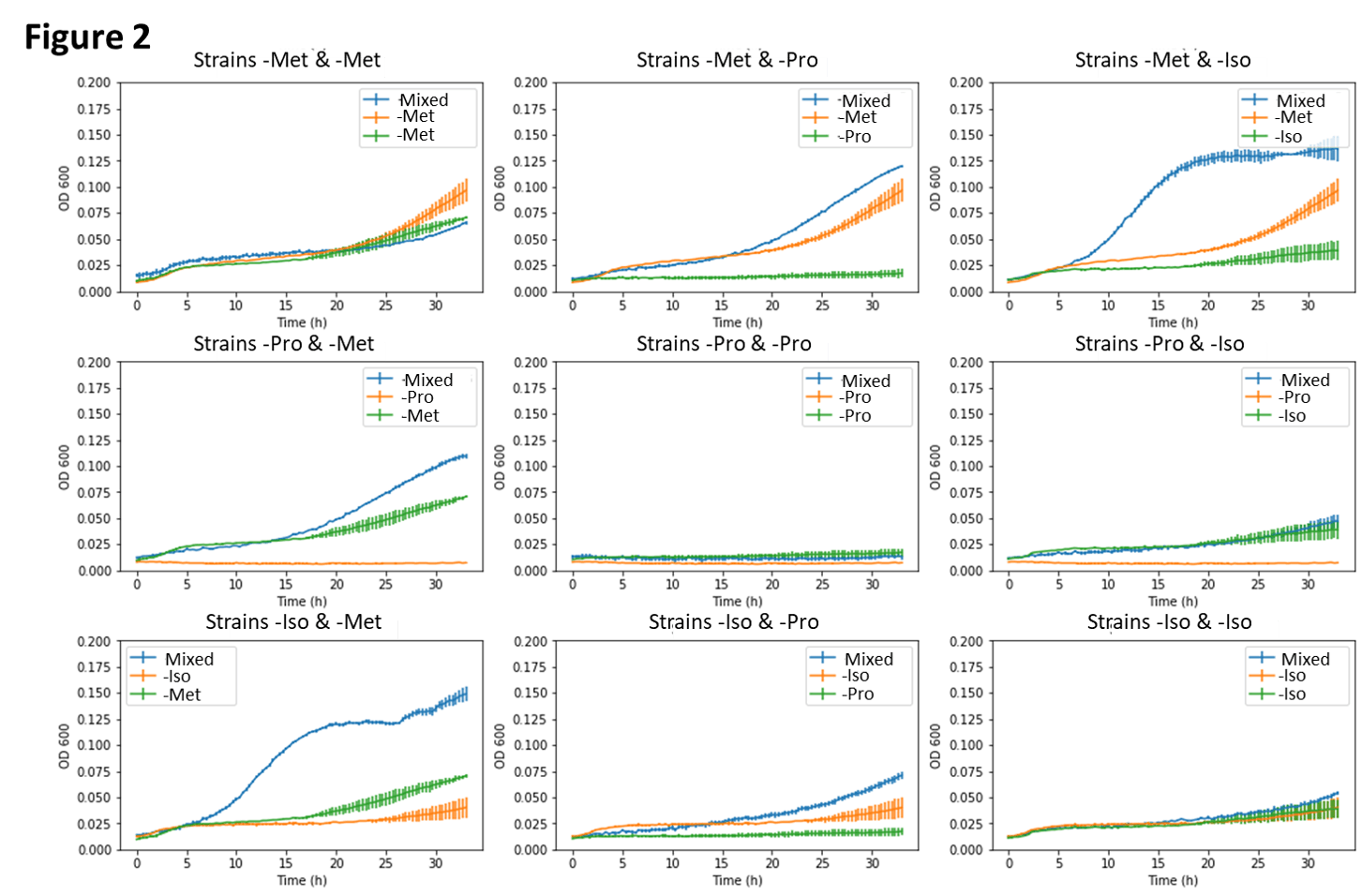
建立营养缺陷型共生培养物
在验证了我们的营养缺陷型菌株的功能特性后,我们接下来将成对的菌株混合在一起,以确定在其他情况下无法存活的菌株在混合时是否可以通过互惠相互作用恢复彼此的生长能力。我们发现这些实验对起始细胞数量、生长培养基进入最小培养基共培养室的残留以及每个营养缺陷型的生存能力差异非常敏感。然而,我们能够在某些营养缺陷型对之间建立强烈的互惠互作,包括 - 蛋氨酸和 - 异亮氨酸对的生长,以及 - 异亮氨酸和 - 异亮氨酸等相同背景的营养缺陷型不生长(图 2)。
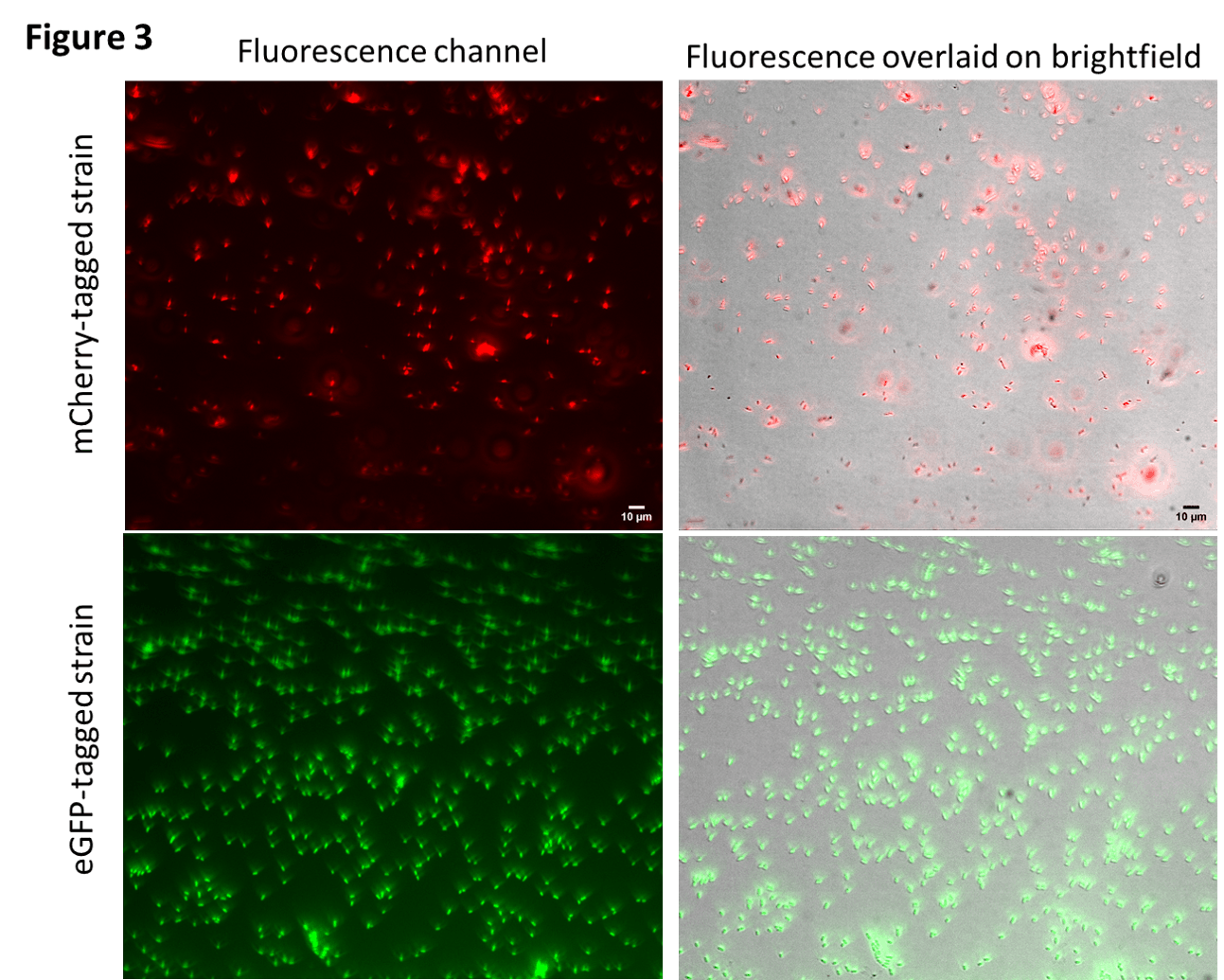
测量基因组编码的荧光标签
为了区分我们正在共同培养的菌株,我们选择了在基因组中集成了不同荧光蛋白标记的兼容菌株(mCherry 用于红色荧光,eGFP 用于绿色荧光)。我们将这些菌株的载玻片和阴性对照(非荧光)和阳性对照(已知荧光)细菌以不同密度放在落射荧光显微镜下。我们发现稀释密度会导致分散的单细胞(与团块或堆叠相反)和视野中的大量细胞(与细胞太少无法分析的稀疏视图相反)。校准阳性对照的显微镜设置后,我们在所有 eGFP 和 mCherry 标记的菌株上看到了清晰的荧光信号。然而,我们也经历了极快的光漂白,信号在照明后几秒钟内消失到无法检测到的水平。尽管我们测试的几种抗褪色封片剂都不能解决这个问题,但我们能够将荧光寿命延长数秒,足以对细胞进行仔细成像(图 3)。
验证纳米体粘附菌株
我们建立了一个协议,用于将我们的粘附质粒引入我们许多不同的标记和营养缺陷型大肠杆菌背景,而无需通常的细菌转化第一步,即首先使细胞具有能力。我们基于电穿孔的方案在从众多菌株中制造转基因细菌方面节省了大量时间,并且具有避免使用冷藏离心机等设备和使用像我们这样的许多社区实验室空间无法使用的快速冷冻的额外好处。
使用此协议,我们生成了带有粘附质粒和荧光标签的菌株,以及一小部分带有粘附质粒的营养缺陷型。在转化之前,我们对所有粘附质粒进行了 Sanger 测序,以验证每个构建体与预期序列完全匹配。这一点特别重要,因为一些肽只有 4 个氨基酸长,否则很难检测到错误。
我们已经开始测试用于编程物理交互的一组表面展示的纳米抗体和抗原对。通过简单的结块测定,我们可以观察到细菌与结合的纳米抗体-抗原组混合的培养物的浊度略有变化,这是由于结合细胞聚集成可以沉淀的致密团块。我们已经看到了三个粘附对中的一个的令人鼓舞的初步结果(图 4A)。在纳米抗体表达细胞和成对的抗原表达细胞的混合物的三个重复中的两个,在第一个小时内出现了显着的结块,而没有纳米抗体匹配的对照没有显着结合(图 4B)。
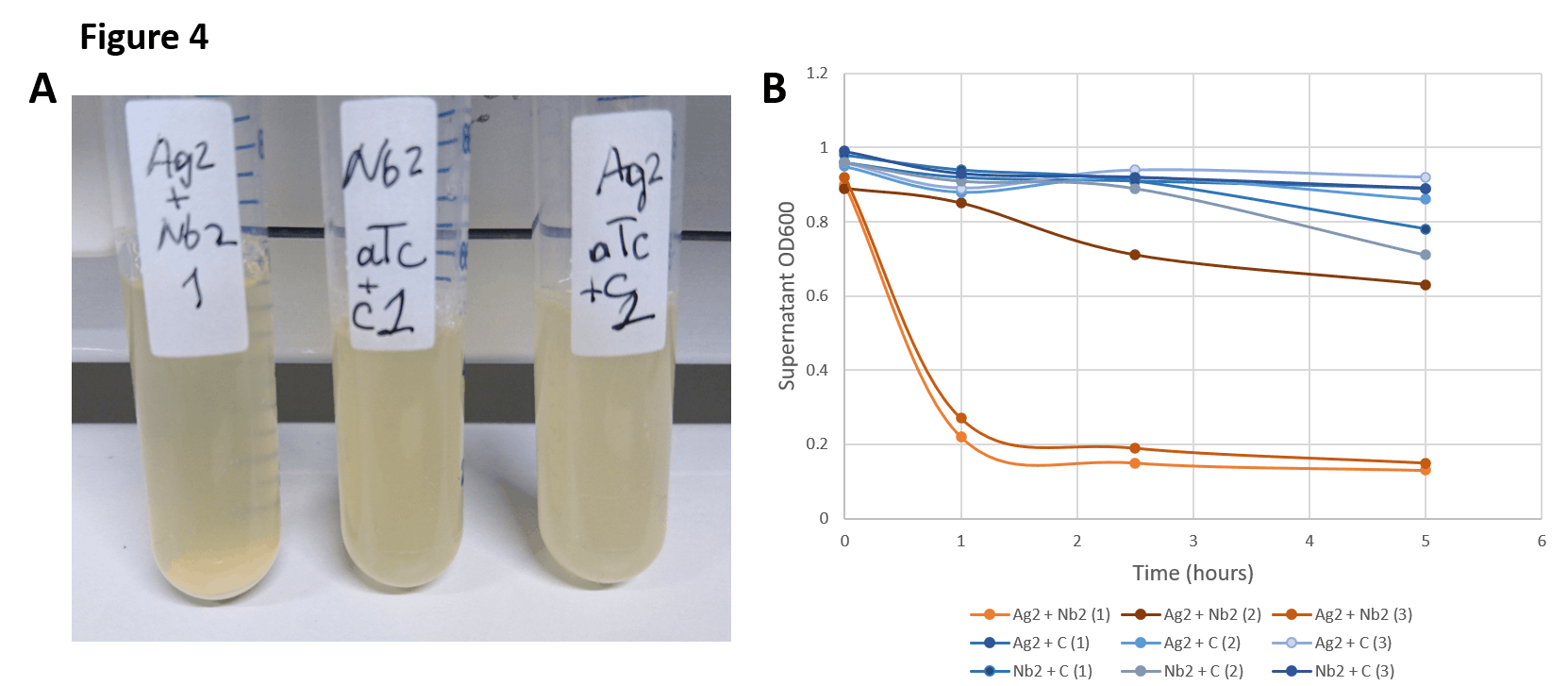
与毒素/抗毒素系统的拮抗相互作用
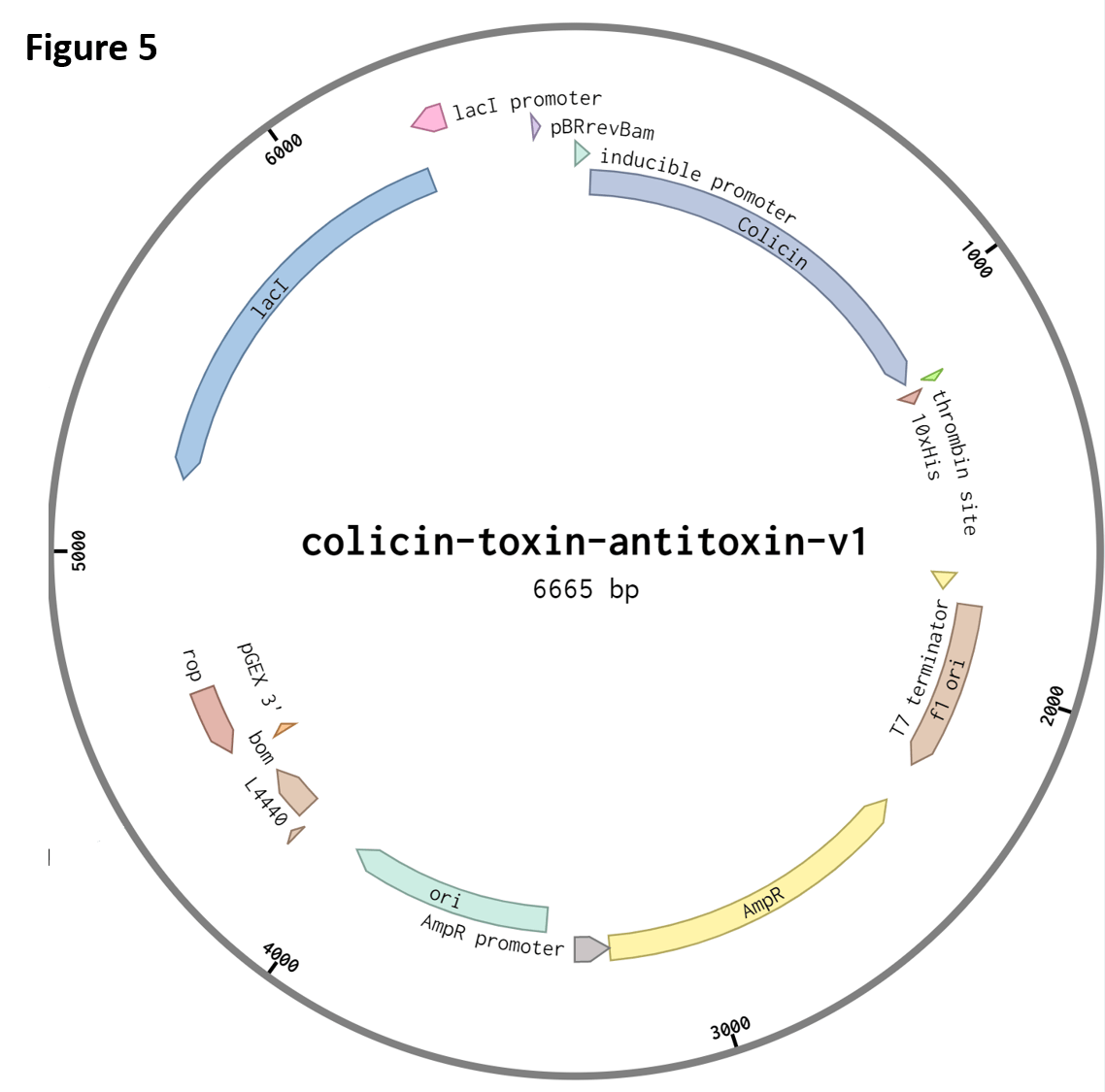
我们还设计了一种质粒,用于产生分泌毒素的细菌,它们自身通过抗毒素的内部表达对其免疫(图 5)。我们为这种毒素-抗毒素质粒选择了大肠杆菌素 E1,并在可诱导的阿拉伯糖启动子的调控下能够滴定毒素水平,从而可以发生有趣的生态相互作用,例如 amensalism,而不会强到没有抗毒素的细胞不能成长和互动。到目前为止,我们已经提取了最终质粒组装所需的一半部分,并将使用我们已建立的生长测定法克隆和测试质粒。
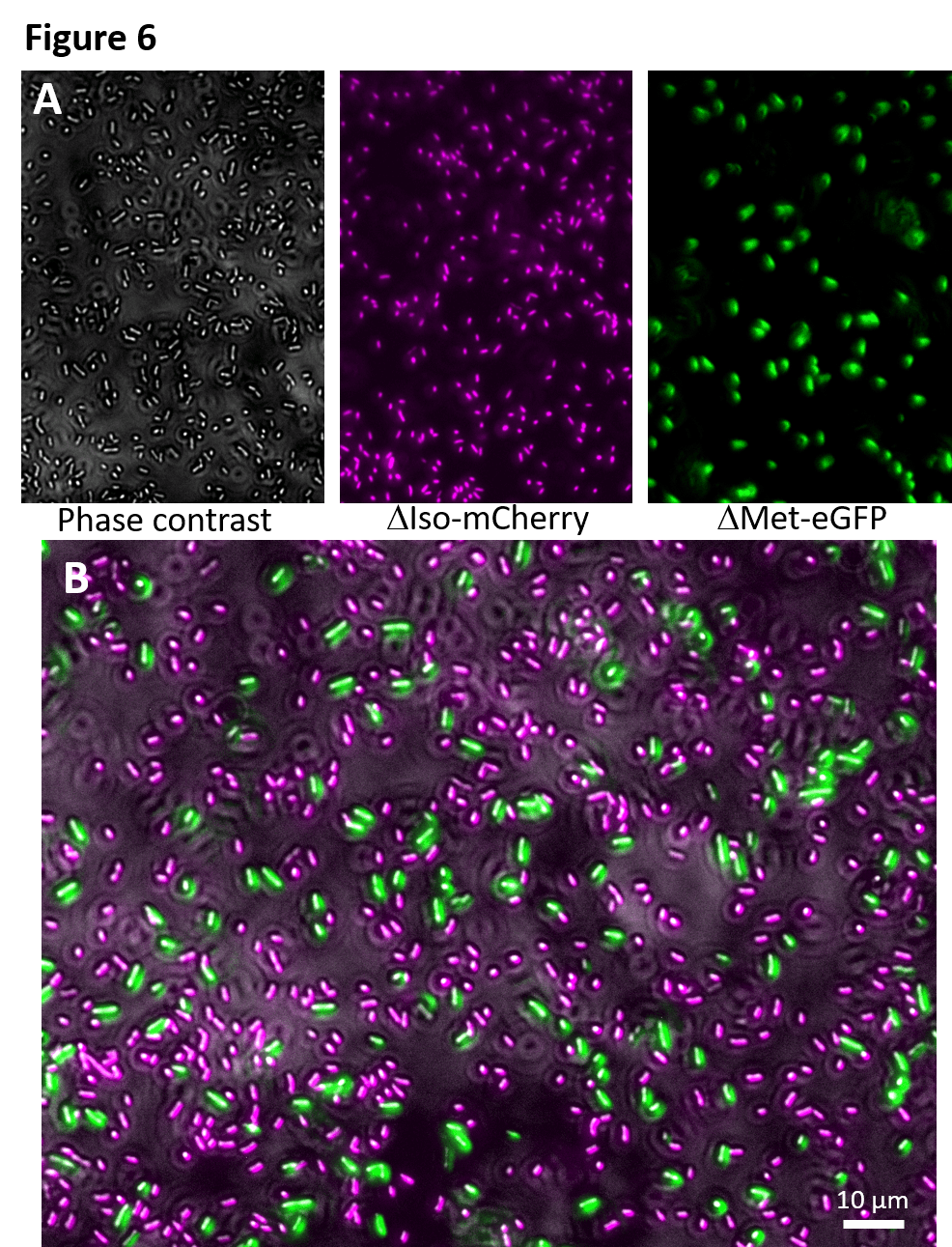
显微镜下的共培养表型
在我们准备和验证了大部分基础菌株后,我们开始描述相互作用细胞的共培养物如何在微观水平上出现。作为第一个例子,我们采用相互依赖的营养缺陷型突变体,让它们一起建立稳定的生长。一个突变体我们标记为绿色 (eGFP),另一个标记为红色 (mCherry)。将细胞从我们的培养板转移到显微镜载玻片上后,我们可以在广角荧光显微镜下观察不同的细胞群(图 6A)。可以从我们的共培养图像中提取大量表型,例如细胞邻近性、比率和定位(图 6B)。虽然我们还没有开始全面探索这些对我们成像数据的潜在分析,但我们打算研究量化共培养图像的方法。
如何使用我们的微生物工具包
对于有兴趣尝试我们的工具包的最终用户,我们在此处提供了一个示例来说明该工具包的工作原理以及我们提供的相应协议和资源。在其最终形式中,用户将收到我们为工具包中的每种大肠杆菌菌株准备的冷冻甘油原液等分试样。这些菌株包括具有营养缺陷型突变、诱导毒素/抗毒素质粒以及表面展示的纳米抗体和抗原的不同组合的细菌。每个菌株在其基因组中都有一个绿色或红色荧光标记,以便在实验中轻松识别。
通过将两种菌株混合在一起,用户可以建立一个明确的双细胞类型群落。我们的协议涵盖了将细菌种群培养成培养物所需的所有基本必要信息,以及在微量滴定板上混合菌株以进行基于平板的实验的方法。遵循这些协议,即使对于营养缺陷型突变体,我们也能够可靠地获得适当的生长行为。重要的是,这些准则允许观察到互利共生的相互作用。
还提供了使用我们的表面粘附蛋白诱导细胞聚集的方案。一旦我们的毒素/抗毒素系统完成,我们还将获得校准诱导水平以获得稳定的拮抗相互作用的说明。我们还有一个使用荧光标签监测共培养细菌生长的程序,这对生态相互作用特别有用。用于分析增长数据的 Python 代码与我们的协议一起提供。
这些协议中的任何一个都可以用来单独描述给定的共文化。我们还有准备细胞样本以在显微镜下观察的方案,我们希望这将成为用户在微生物群落复杂性不断增加的水平上进行探索的特别有趣的观察来源。
外表
我们希望尽快完成组装构成我们交互工具箱的粘附菌株和突变菌株。我们正在努力优化的协议将是该工具包的另一个关键组成部分,因此其他人可以轻松建立适当的培养条件来观察有趣的生物学效应。
我们的工具包有许多潜在的应用。首先,它可能会用作一种教育资源,以通过易于查看的读数显示简单设置中的微生物相互作用。其次,它们可以成为在还原论框架下研究微生物相互作用的研究工具。复杂细胞自组装的可能性特别有趣,并且有来自其他人的数据支持其合理性。第三,混合细胞群之间的稳定相互作用可用于优化生物反应器,超出单一培养所能达到的范围。最后,能够对这些相互作用进行编程可以将细胞转变为用于生产图案化生物材料的支架。通过我们作为 Biomaker Challenge 的一部分的工作以及我们作为剑桥大学合成生物学协会的学生的项目,
参考
玻璃 DS,Riedel-kruse IH。用于编程多细胞形态和模式的合成细菌细胞-细胞粘附工具箱。细胞。2018;174(3):649-658.e16。
杰克斯堪萨斯州。大肠杆菌素 E1 TolC 盒:鉴定大肠杆菌素 E1 细胞毒性和 TolC 结合所需的结构域。J细菌学。2017;199(1)
Marchal M、Goldschmidt F、Derksen-müller SN、Panke S、Ackermann M、Johnson DR。被动的共生相互作用促进了微生物种群内空间结构的进化。BMC 进化生物学。2017;17(1):106。
Kong W, Meldgin DR, Collins JJ, Lu T. 设计具有明确社会互动的微生物群落。自然化学生物学。2018;14(8):821-829。
声明:本文内容及配图由入驻作者撰写或者入驻合作网站授权转载。文章观点仅代表作者本人,不代表电子发烧友网立场。文章及其配图仅供工程师学习之用,如有内容侵权或者其他违规问题,请联系本站处理。 举报投诉
- 相关下载
- 相关文章







